

背景簡介
microRNA(miRNA)是通過(guò)轉錄後(hòu)水平調控的靶基因(mRNA)的表達來實現其重要生物學(xué)功能(néng)。在生物體内,miRNA除了抑制mRNA的翻譯外,也會(huì)誘導mRNA被剪切降解,從而調控基因表達,發(fā)揮生物學(xué)功能(néng)。相比于動物,在植物中miRNA誘導基因剪切降解的方式會(huì)更爲普遍。miRNA剪切靶基因會(huì)産生二個片段,5‘剪切片段和3’剪切片段,其中3‘剪切片段,包含有自由的5’單磷酸和3‘polyA尾巴,可被磁珠富集,并可在RNA連接酶的作用下連接接頭,連接産物經(jīng)擴增可用于下遊的高通量測序。降解組測序正是對(duì)mRNA的降解片段進(jìn)行測序,進(jìn)而可靠地大規模實驗鑒定miRNA靶基因的一種(zhǒng)高效工具.
技術優勢
用戶文章數量最多:用戶發(fā)表降解組文章約占全球降解組實驗性文章總數1/3,國(guó)内最多。
業内領先的文庫構建:樣本起(qǐ)始量更低,建庫步驟更少、測序讀長(cháng)更長(cháng),更真實反映樣本降解組的豐度,顯著提高數據準确性
數據分析軟件優:使用自主開(kāi)發(fā)的數據分析軟件ACGT301-DEG101,并結合CleaveLand,分析可靠性經(jīng)過(guò)數千個實驗項目檢驗
項目經(jīng)驗豐富:國(guó)内最早開(kāi)展降解組測序服務,成(chéng)熟的研究組合方案,幫助用戶研究成(chéng)果快速發(fā)表。
技術路線
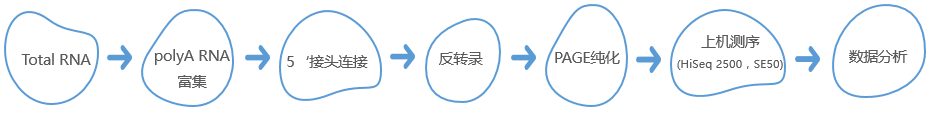
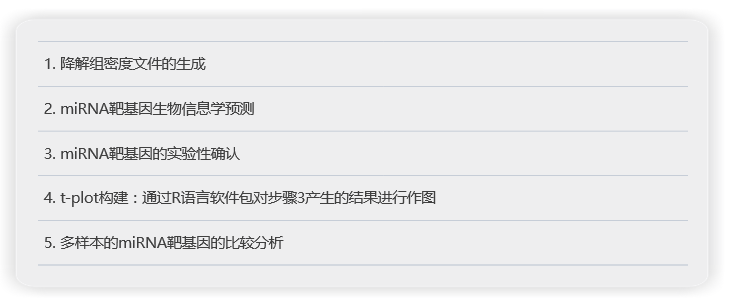
分析内容
樣本類型
細胞,組織,總RNA等
建議總RNA起(qǐ)始量:50 μg,最低25 μg,濃度≥400 ng/μL
近期用戶文章
1. Li H, Peng T, Wang Q, Wu Y, Chang J, Zhang M, Tang G, Li C. (2017) Development of Incompletely Fused Carpels in Maize Ovary Revealed by miRNA, Target Gene and Phytohormone Analysis. Frontiers in Plant Science 8(1), 463.
2. Wang Q, Li T, Xu K, Zhang W, Wang X, Quan J, Jin W, Zhang M, Fan G, Wang M. (2016) The tRNA-Derived Small RNAs Regulate Gene Expression through Triggering Sequence-Specific Degradation of Target Transcripts in the Oomycete Pathogen Phytophthora sojae. Frontiers in Plant Science 7,
3. Tang F, Wei H, Zhao S, Wang L, Zheng H, Lu M. (2016) Identification of microRNAs Involved in Regeneration of the Secondary Vascular System in Populus tomentosa Carr. Frontiers in Plant Science 7(1),
4. Gao F, Wang N, Li H, Liu J, Fu C, Xiao Z, Wei C, Lu X, Feng J, Zhou Y. (2016) Identification of drought-responsive microRNAs and their targets in Ammopiptanthus mongolicus by using high-throughput sequencing. Scientific Reports 6(1), 34601.
5. Zhou R, Wang Q, Jiang F, Cao X, Sun M, Liu M, Wu Z. (2016) Identification of miRNAs and their targets in wild tomato at moderately and acutely elevated temperatures by high-throughput sequencing and degradome analysis. Scientific Reports 6(1), 33777.
6. Zhang J, Huang M, Liang J, Pan Y, Cheng L, Wu J, Tong Z. (2016) Genome-wide mining for microRNAs and their targets in Betula luminifera using high-throughput sequencing and degradome analyses. T
Q1:降解組測序所适用的物種(zhǒng)?
A:相對(duì)于動物,植物更适用于運用降解組測序研究miRNA的靶基因。而相對(duì)于轉錄組信息并不全的物種(zhǒng)來說(shuō),模式生物或者有詳細轉錄組信息的物種(zhǒng)更适用于運用降解組測序研究miRNA的靶基因。
Q2:降解組測序實驗是否保證測序實驗結果的數據量?
A:聯川公司承諾每一個降解組測序實驗均保證測序數據量達到500萬以上。
Q3:降解組測序除了能(néng)檢測miRNA的靶基因外還(hái)能(néng)做哪些研究?
A:首先我們需要了解降解組測序最大的一個作用就(jiù)是尋找miRNA的靶基因,當然随著(zhe)研究人員對(duì)降解組數據進(jìn)行深入的分析發(fā)現其也能(néng)用于研究miRNA的自我調控、ta-siRNA、前體序列的加工以及用于檢測新的miRNA等。本公司目前所提供給客戶的僅爲miRNA的靶基因信息,對(duì)于更進(jìn)一步的數據分析,您可與本公司技術客服溝通定制。
研究背景
種(zhǒng)子休眠,包括初生休眠和次生休眠,不僅是植物應對(duì)不利環境的一種(zhǒng)适應對(duì)策,而且可以阻止作物的成(chéng)熟種(zhǒng)子在收獲前萌發(fā),從而有效避免減産。另一方面(miàn),打破種(zhǒng)子休眠則可以促進(jìn)種(zhǒng)子萌發(fā),從而在作物種(zhǒng)植和林業育苗中實現整齊出苗。因此,了解種(zhǒng)子的休眠特性及其調控機制,在農林業生産實踐中具有極其重要的意義,然而這(zhè)方面(miàn)的研究卻鮮爲人知。
研究結果
本項研究以我國(guó)重要經(jīng)濟林樹種(zhǒng)杉木爲研究對(duì)象,綜合使用透射電子顯微技術、轉錄組測序、降解組測序、 ACGT101-miR分析和高效液相色譜—質譜等多種(zhǒng)技術,研究杉木新成(chéng)熟種(zhǒng)子、 12d低溫層積處理種(zhǒng)子、 35℃儲存40d種(zhǒng)子休眠釋放和誘導過(guò)程中,細胞學(xué)、基因表達和激素水平等方面(miàn)的變化,系統闡釋了杉木種(zhǒng)子的休眠循環過(guò)程及其調控機制。光學(xué)顯微鏡和透射電子顯微鏡顯示,種(zhǒng)子初生休眠釋放期,蛋白體在胚胎細胞中合并,而在次生休眠誘導期間分離。轉錄組結果分析表明,初生休眠釋放期間,負調控GA敏感性的基因顯著下調表達;次生休眠誘導期間,正調控ABA生物合成(chéng)的基因顯著上調表達。在種(zhǒng)子休眠釋放和誘導期間,細胞學(xué)和基因表達的可逆變化與ABA/GA平衡有關。此外,初生休眠期間,mRNA降解可以作爲關鍵性的轉錄後(hòu)調節器發(fā)揮功能(néng)。一些miRNA通過(guò)調控激素信号關鍵基因參與種(zhǒng)子生理休眠過(guò)程。
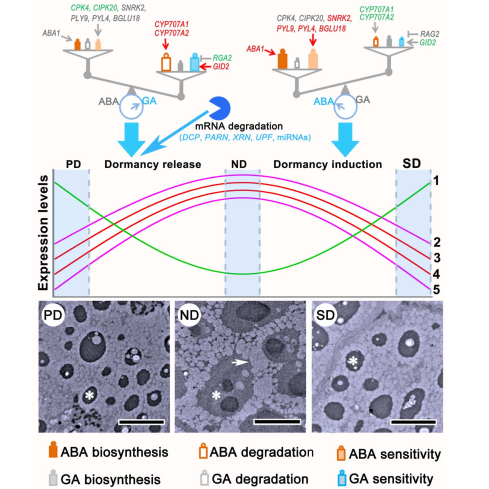
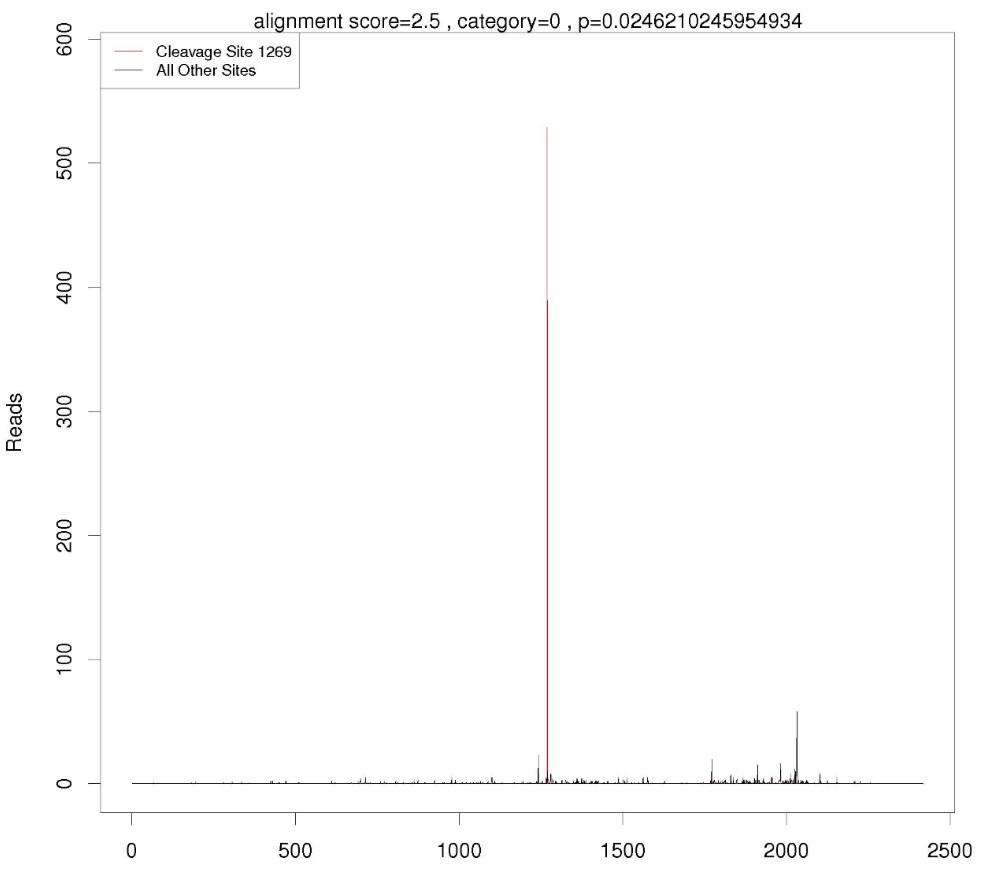
圖 杉木種(zhǒng)子生理休眠的調控機制和初生、次生休眠間的差異
參考文獻
Cao DC, et al. (2016) Transcriptome and Degradome Sequencing Reveals Dormancy Mechanisms of Cunninghamia lanceolata Seeds. Plant Physiology.DOI:10.1104/pp.16.00384
GO富集性柱狀圖在所有的選定的miRNA靶基因中,將(jiāng)這(zhè)些基因對(duì)應的GO注釋按照Molecular Function、Biological Process和Cellar Component分爲三類, 并且將(jiāng)每一類中的GO功能(néng)按照注釋到的靶基因個數從高到低排序,并 進(jìn)行作圖,橫坐标是對(duì)GO的分類,縱坐标是靶基因所占的百分比,可 以直觀地看出注釋到同一個GO的靶基因數目所占的百分比。
GO富集性散點圖GO的基本單位是term(詞條、節點),每個term都(dōu)對(duì)應一個屬性。GO功能(néng)顯著性富集分析首先把所有顯著性差異表達基因向(xiàng)Gene Ontology數據庫的各term映射,計算每個term的基因數目,然後(hòu)應用超幾何檢驗,找出與整個基因組背景相比,在顯著性差異表達基因中顯著富集的GO條目。聯川生物采用ggplot2對(duì)GO富集分析結果以散點圖展示:Rich factor表示位于該GO的差異基因個數/位于該GO的總基因數,P值越小,GO富集程度越高。
KEGG通路圖Pathway通路圖展示與說(shuō)明:紅色代表注釋到某個ko節點且上調的顯著差異表達基因,藍色或紫色代表注釋到某個ko節點且下調的顯著差異表達基因。方框内的4位數字表示各種(zhǒng)酶的EC編号;空心圓圈表示小分子化合物;實心箭頭表示生化反應的方向(xiàng);虛線箭頭連接其他的相關代謝途徑。下圖僅爲報告中的展示圖片ko04310。
KEGG通路分類圖KEGG PATHWAY數據庫是一個手工畫的代謝通路的集合,包含以下幾方面(miàn)的分 子間相互作用和反應網絡:1.新陳代謝、2.遺傳信息加工、3.環境信息加工、4.細胞過(guò)程、5.生物體系統、6.人類疾病、7.藥物開(kāi)發(fā)。圖中對(duì)KEGG pathway進(jìn)行了分類,并且顯示每條通路上的基因數目.
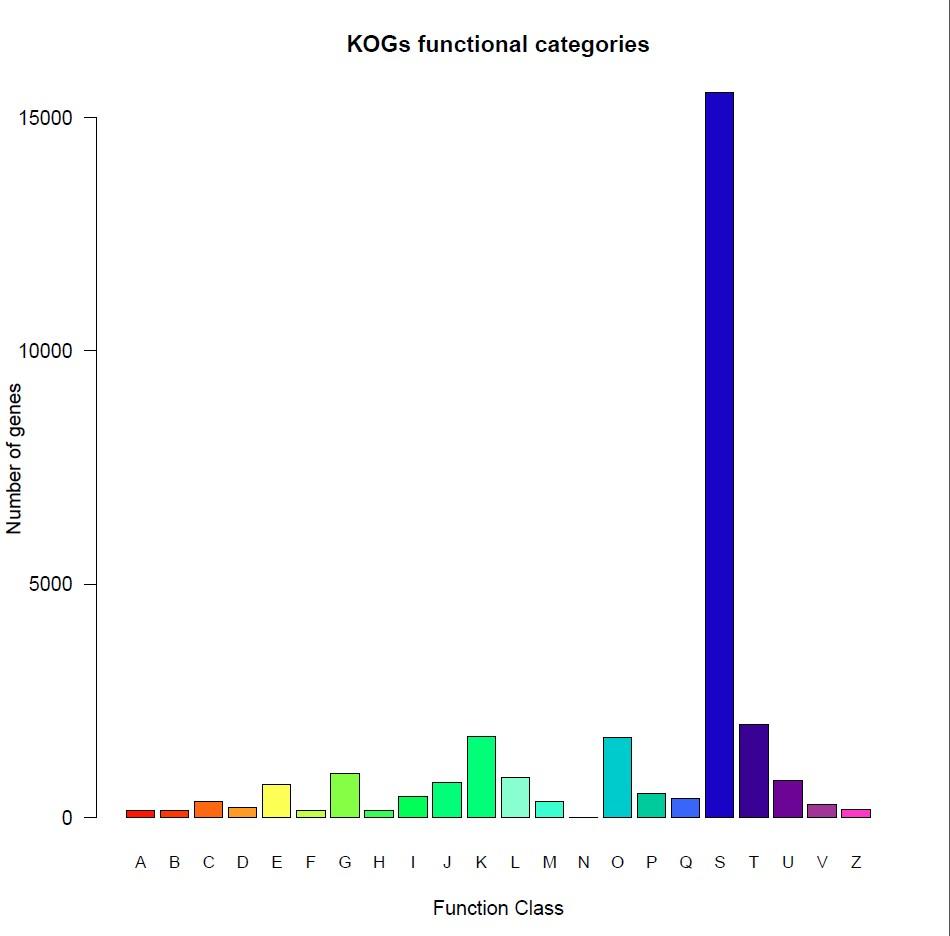
KOG分類圖KOG 是 Clusters of orthologous groups for eukaryotic complete genomes(真核生物蛋白相鄰類的聚簇)的縮寫。構成(chéng)每個 KOG 的蛋白都(dōu)是被假定爲來自于一個祖先蛋白,并且因此或者是 orthologs 或者是 paralogs。Orthologs是指來自于不同物種(zhǒng)的由垂直家系(物種(zhǒng)形成(chéng))進(jìn)化而來的蛋白,并且典型的保留與原始蛋白有相同的功能(néng)。Paralogs 是那些在一定物種(zhǒng)中的來源于基因複制的蛋白,可能(néng)會(huì)進(jìn)化出新的與原來有關的功能(néng)。對(duì)基因進(jìn)行 KOG 功能(néng)分類預測:共有 25 個 KOG 分類。
targets-plot,靶基因鑒定信息圖將(jiāng)降解組序列文件和mRNA序列文件進(jìn)行配對(duì)并生成(chéng)降解組密度文件(degradome density file),文件内包括mRNA序列編号、長(cháng)度、配對(duì)位點、reads數和RPM并給出降解組的峰值分類,然後(hòu)對(duì)産生的預測結果進(jìn)行作圖,以圖形的模式更直觀的展示所檢測到的miRNA相對(duì)的靶基因。