

背景簡介
細菌基因組重測序是指對(duì)基因組序列已知的細菌個體進(jìn)行基因組測序,通過(guò)與已知的參考基因組比對(duì),獲得該細菌個體或者群體的差異的測序方法,這(zhè)些差異主要包括SNP,InDel和SV。目前微生物基因組重測序被廣泛應用于病原微生物的檢測及鑒定、病原菌演變及起(qǐ)源、緻病菌種(zhǒng)群結構及種(zhǒng)群結構的進(jìn)化等衆多方面(miàn)。
技術優勢
細菌基因組重測序能(néng)夠發(fā)現未知的遺傳變異信息。
憑借二代測序,細菌基因組重測序獲得數據通量更高,速度更快,成(chéng)本更低。
細菌基因組重測序可以更全面(miàn)的檢測SNP、InDel、SV等多種(zhǒng)遺傳變異類型。
技術路線
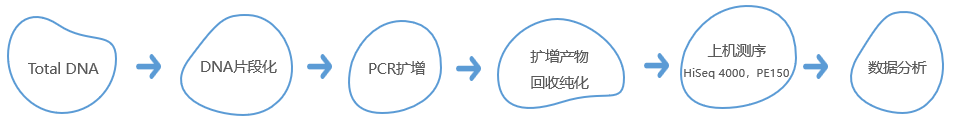
分析内容
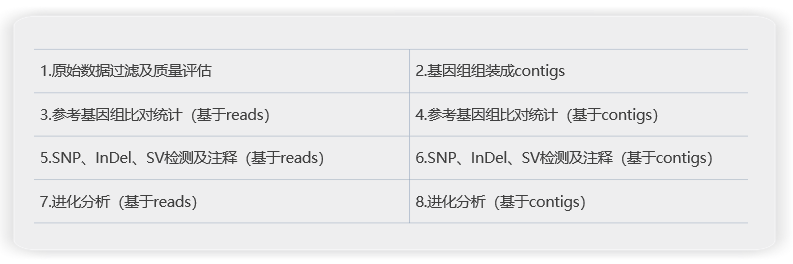
樣本類型
細菌菌體,環境樣品,總DNA等
建議總DNA起(qǐ)始量:≥5 μg,最低起(qǐ)始量:0.5ug,濃度≥30 ng/μl(Qubit定量)
Q1:細菌基因組完成(chéng)圖是否可以組裝出質粒?
A:細菌細胞内一般會(huì)存在部分質粒,細菌基因組完成(chéng)圖可以組裝出部分質粒信息,但是由于建庫的長(cháng)度以及質粒的數量限制,不保證完全組裝出所有質粒。
Q 2:細菌基因組測序中比較基因組分析是什麼(me)?
A:細菌基因組比較基因組分析,主要是分析有親緣關系的細菌基因之間的差别,通過(guò)比較基因組分析親緣遠近關系,特有和共有基因以及共線性分析等,找到差異基因,對(duì)差異基因進(jìn)行功能(néng)注釋,解析近緣關系細菌間生理或形态差别的原因。
Q3:在重測序中,爲什麼(me)隻能(néng)得到插入/缺失了堿基的數目,卻得不到插入/缺失的具體位置和序列信息?如何能(néng)夠獲得具體的序列信息呢?
A:在重測序中,SV檢測分析是可以得到樣本相對(duì)于參考基因組的一個大概的DEL序列的。由于重測序中隻是對(duì)于文庫片段的兩(liǎng)端進(jìn)行測序,所以中間的INS序列暫時(shí)無法檢測到;理論上,可以對(duì)插入位置附件設計引物,通過(guò)PCR擴增出具體的序列,此外,也可以通過(guò)局部組裝附近的reads來獲取中間的序列信息(主要取決于局部組裝的效果)。
腸外緻病性大腸杆菌的大規模基因組測序
研究背景
大腸杆菌(E. coli)爲埃希氏菌屬(Escherichia)代表菌。一般多爲條件緻病菌,某些血清型菌株的緻病性強,嚴重腹瀉和敗血症,統稱緻病性大腸杆菌。其中,緻病性大腸杆菌的抗生素耐藥性,是相關醫療實踐中的一個重要問題。
方法流程
取材:1. 5個月時(shí)間從277個病人尿液中分離出288株尿源E.coli ;2. 3年時(shí)間從47名病人血液中分離培養出92株血源E.coli
建庫:構建小片段文庫
測序:Illumina HiSeq 2000
分析:1. 基因組組裝;2. Core-pan基因分析;3. 全基因組關聯分析
研究結果
1. Core-pan基因分析
通過(guò)Core-pan基因分析發(fā)現,ExPEC E.coli具有高度遺傳異質性,并區分爲不同種(zhǒng)系。不同緻病因素及抗生素抗性表型對(duì)應人體不同部位的感染性。
2. 緻病性研究
高分辨率的分子流行病學(xué)研究顯示,經(jīng)由血液傳播的亞種(zhǒng)比例較低,僅占全部亞種(zhǒng)的28%。
3. 抗性基因挖掘
全基因組關聯分析發(fā)現部分抗生素抗性相關的基因,并成(chéng)功驗證了部分已知抗性的基因。
研究結論
本文嘗試將(jiāng)大量高通量數據應用到醫療機構,并從中尋找到菌種(zhǒng)分類、進(jìn)化及抗生素抗性等重要基因信息的獲得途徑,爲後(hòu)續疾病相關大規模微生物全基因組測序提供了範例。
SNP分析SNP全稱Single Nucleotide Polymorphisms,是指在基因組上單個核苷酸 的變異,形成(chéng)的遺傳标記,其數量很多,多态性豐富。基因組上單個核 苷酸的變異包括置換,缺失和插入。采用FreeBayes對(duì)各樣品比對(duì)結果進(jìn)行個體SNP的檢測。
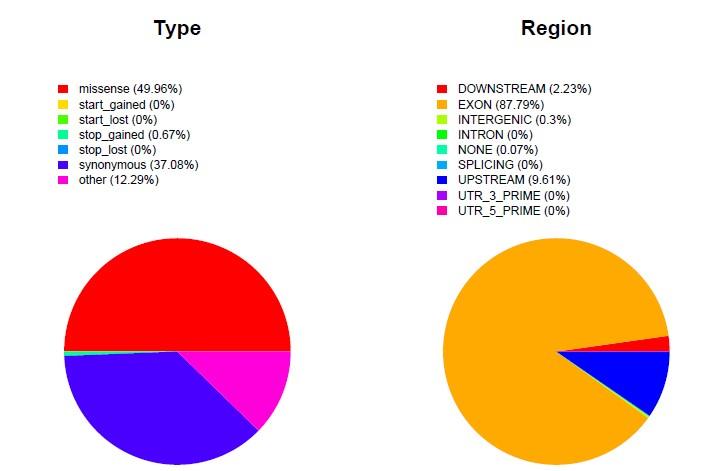
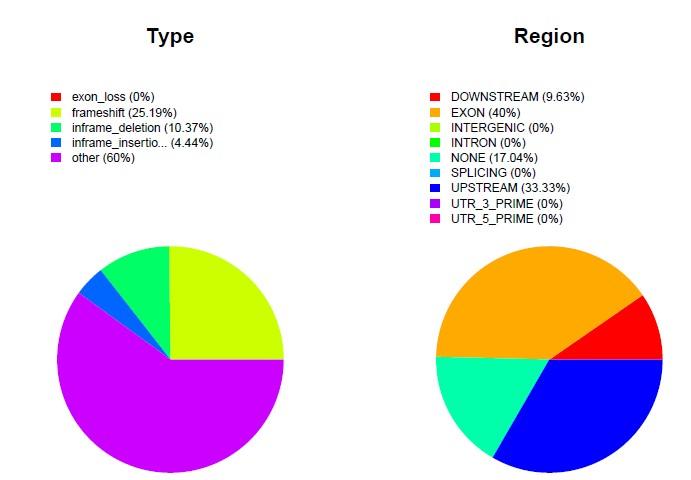
InDel分析InDel (Insertion-Deletion) 是指相對(duì)于參考基因組,樣本中發(fā)生的小片段的 插入缺失,該插入缺失可能(néng)含一個或多個堿基。根據InDel在基因組中的位 置,可以分爲編碼區序列的InDel和非編碼區序列的InDel。編碼序列中的 InDel發(fā)生與編碼蛋白質的功能(néng)和氨基酸位點在結構和功能(néng)上的重要性有關。 采用FreeBayes對(duì)各樣品比對(duì)結果進(jìn)行個體InDel的檢測。
SNP進(jìn)化樹分析在生物學(xué)中,進(jìn)化分析是指根據遺傳或者表型的差異研究推斷不同物種(zhǒng)或者個體間的進(jìn)化關系的一門學(xué)科。進(jìn)化分析的結果通常用進(jìn)化樹的形式展示,在進(jìn)化樹上每個結點代表一個物種(zhǒng)或個體,那麼(me)兩(liǎng)個結點之間的最短距離就(jiù)表示相應的兩(liǎng)個物種(zhǒng)之間的差異程度。根據每個個體檢測到的SNP位點,過(guò)濾掉彼此距離較近的SNP位點(<20bp) ,因爲這(zhè)些位點可能(néng)是由基因組重組産生,并不能(néng)真正反映物種(zhǒng)間的進(jìn)化關系。將(jiāng)每個個體非重組的SNP位點堿基串聯成(chéng)一條序列,得到個體多序列比對(duì)的結果,最後(hòu)構建最大似然進(jìn)化樹。