常規轉錄組測序由于文庫PCR擴增偏好(hǎo)性,所有序列并不會(huì)被同比例放大,因而造成(chéng)測序定量結果與樣本中轉錄本的原始豐度不一緻,導緻差異基因篩選不準确(圖1)。這(zhè)也是造成(chéng)qPCR驗證測序結果不一緻的主要原因。聯川生物推出的絕對(duì)定量轉錄組測序,采用主流UMI标記技術[1,2,3],通過(guò)UMI标記每一條序列,可以消除PCR擴增偏好(hǎo)對(duì)定量的幹擾,真實反映樣本中轉錄本的表達豐度(圖2)。在文庫PCR擴增和測序過(guò)程中難免會(huì)引入錯誤的堿基,具有相同UMI标記的序列可以基于多序列比對(duì)來糾正PCR擴增和測序過(guò)程中引入的錯誤,确保獲得轉錄本的真實序列(見圖3)。
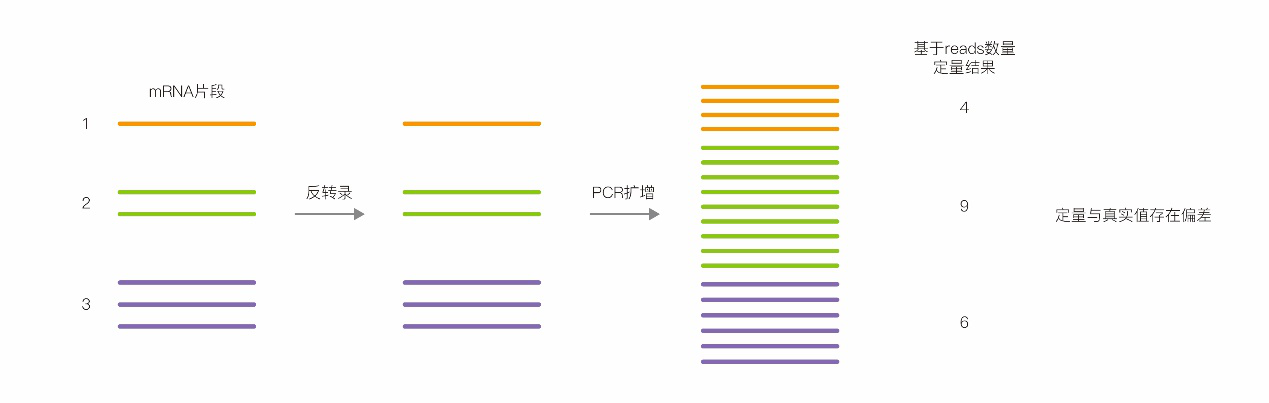
圖1 PCR擴增産生的duplication顯著幹擾準确定量
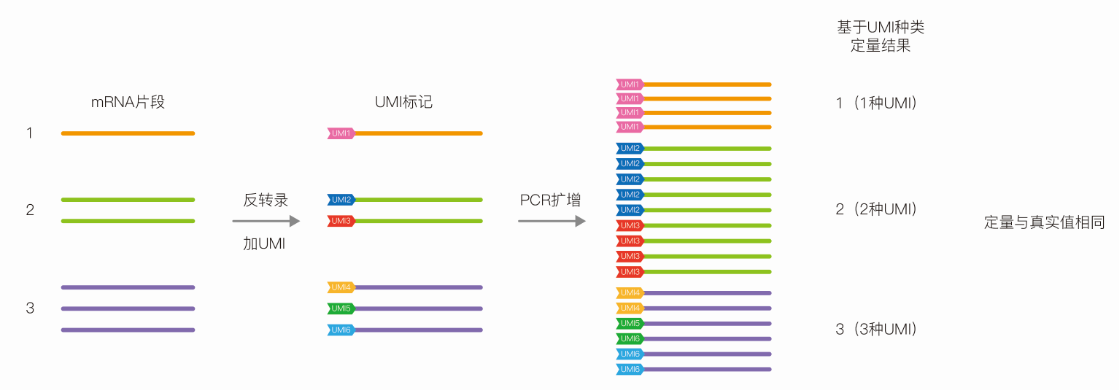
圖2 UMI标記技術消除duplication幹擾
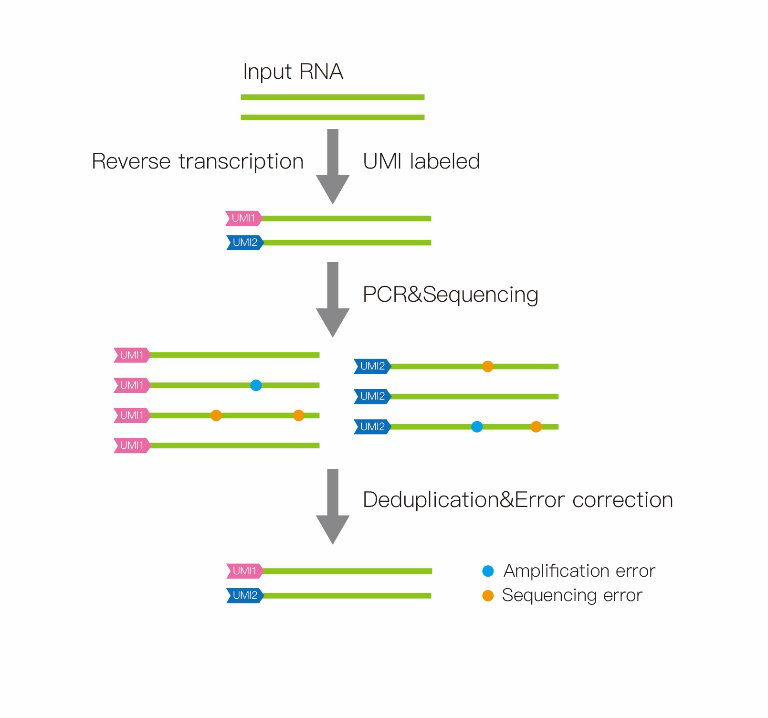
圖3 UMI标記技術糾正序列錯誤
真實定量:采用UMI标記技術,每一條序列都(dōu)被唯一标識,保留序列真重複,去除PCR擴增假重複,真實反映樣本中轉錄本的表達豐度。真實序列:具有相同UMI标記的序列可以基于多序列比對(duì)來糾正PCR擴增和測序過(guò)程中引入的錯誤, 确保獲得轉錄本的真實序列。
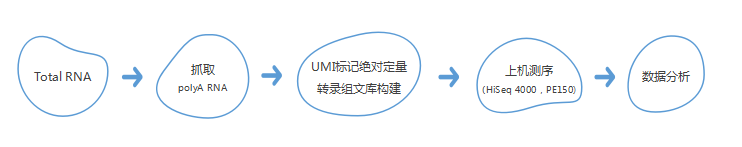
細胞,組織,全血,血清,血漿,總RNA等
建議總RNA起(qǐ)始量:2 μg,最低1 μg,濃度≥50 ng/μL
參考文獻
1.Shiroguchi K, et al. Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single-molecule barcodes. Proc Natl Acad Sci U S A. 2012 Jan 24;109(4):1347-52.2.Kivioja T, et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat Methods. 2011, 9(1):72-43.Saiful Islam, et al. Quantitative single-cell RNA-seq with unique molecular identifiers. Nature Methods 2014, 11:163-166.
A1:主要有這(zhè)6個原因:1)文庫PCR擴增偏好(hǎo),2)你驗證的分子不對(duì),3)用不同批次的樣品做驗證,4)用不同的樣本類型做驗證,5)拿低表達或差異不顯著的基因做驗證,6)樣本比對(duì)關系搞反。查看詳情
A2:使用絕對(duì)定量轉錄組測序,助您篩選出真實的差異表達基因,讓你的時(shí)間不再花在假陽性結果的驗證上,更快地開(kāi)展下遊功能(néng)驗證實驗,快人一步地發(fā)表研究成(chéng)果。
這(zhè)項工作讨論了如何最小化PCR擴增偏好(hǎo)性(就(jiù)是Duplication)對(duì)測序數據,特别是低拷貝序列,定量分析的幹擾。研究團隊開(kāi)發(fā)出了一種(zhǒng)稱爲Digital RNA sequencing的方法:序列在反轉錄後(hòu),加入大量的标簽(barcode),幾乎每個cDNA都(dōu)被唯一的barcode标記,然後(hòu)進(jìn)行PCR擴增獲得轉錄組測序文庫(見下圖)。由于序列是被barcode唯一标記的,計算序列拷貝數時(shí),不再直接統計同一種(zhǒng)reads的數量,而是統計每種(zhǒng)reads有多少個unique的barcode。而具有相同barcode的同一種(zhǒng)reads,無論有多少拷貝數,都(dōu)隻計作一個拷貝,即來自PCR擴增的假重複(Duplication)被有效去除。
可以簡單地認爲,在樣本中cDNA1有3個拷貝,cDNA2有2個拷貝,比例爲3:2,然後(hòu)用大量不同的barcode标記這(zhè)5條序列,每條序列都(dōu)被unique的barcode标記,最後(hòu)進(jìn)行PCR擴增完成(chéng)文庫制備。由于Duplication的存在,在沒(méi)有barcode标記的情況下,cDNA1經(jīng)擴增變成(chéng)了9個拷貝,cDNA2變成(chéng)了12個拷貝,比例變成(chéng)了3:4,而兩(liǎng)者原始的比例是3:2;而标記了barcode的cDNA1和cDNA2,合并具有相同barcode的同種(zhǒng)序列後(hòu),cDNA1和cDNA2仍保持原始的拷貝數和比例,此種(zhǒng)方案下測序定量結果準确反映了樣本中序列的真實豐度和比例。
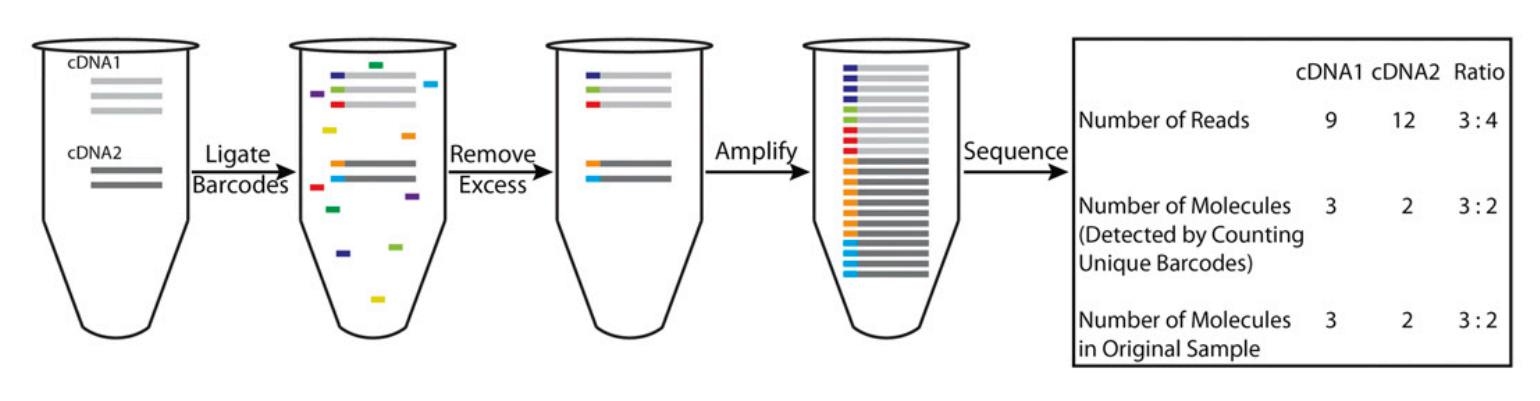
圖 标簽标記序列法去除Duplication的原理
Shiroguchi K, et al. Digital RNA sequencing minimizes sequence-dependent bias and amplification noise with optimized single-molecule barcodes. Proc Natl Acad Sci U S A. 2012 Jan 24;109(4):1347-52.
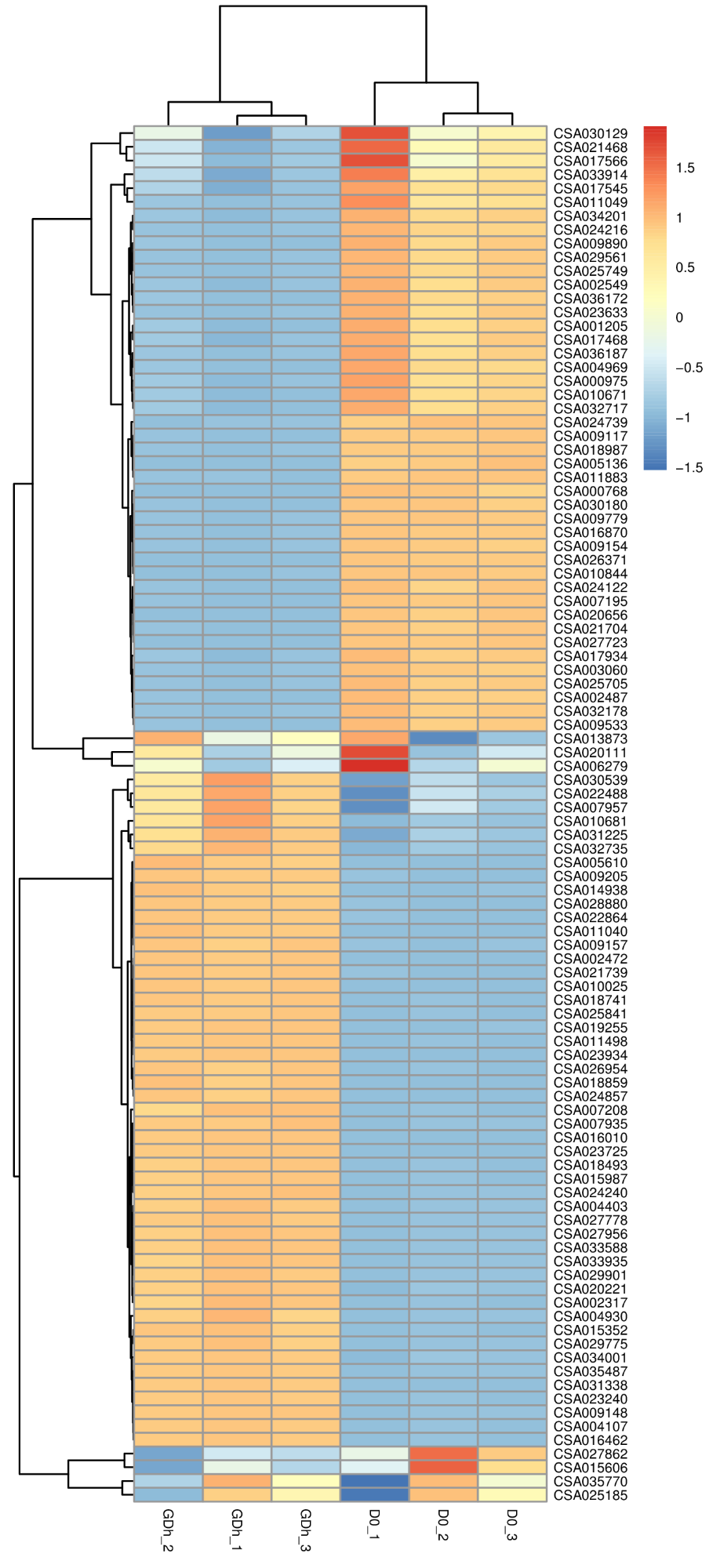
差異基因聚類分析熱圖
用于判斷基因在不同實驗條件下調控模式的聚類模式根據樣品基因表達譜的相近程度,將(jiāng)基因進(jìn)行聚類分析,直觀地展示基因在不同樣品(或是不同處理)中的表達情況,由此獲取生物學(xué)相關信息。
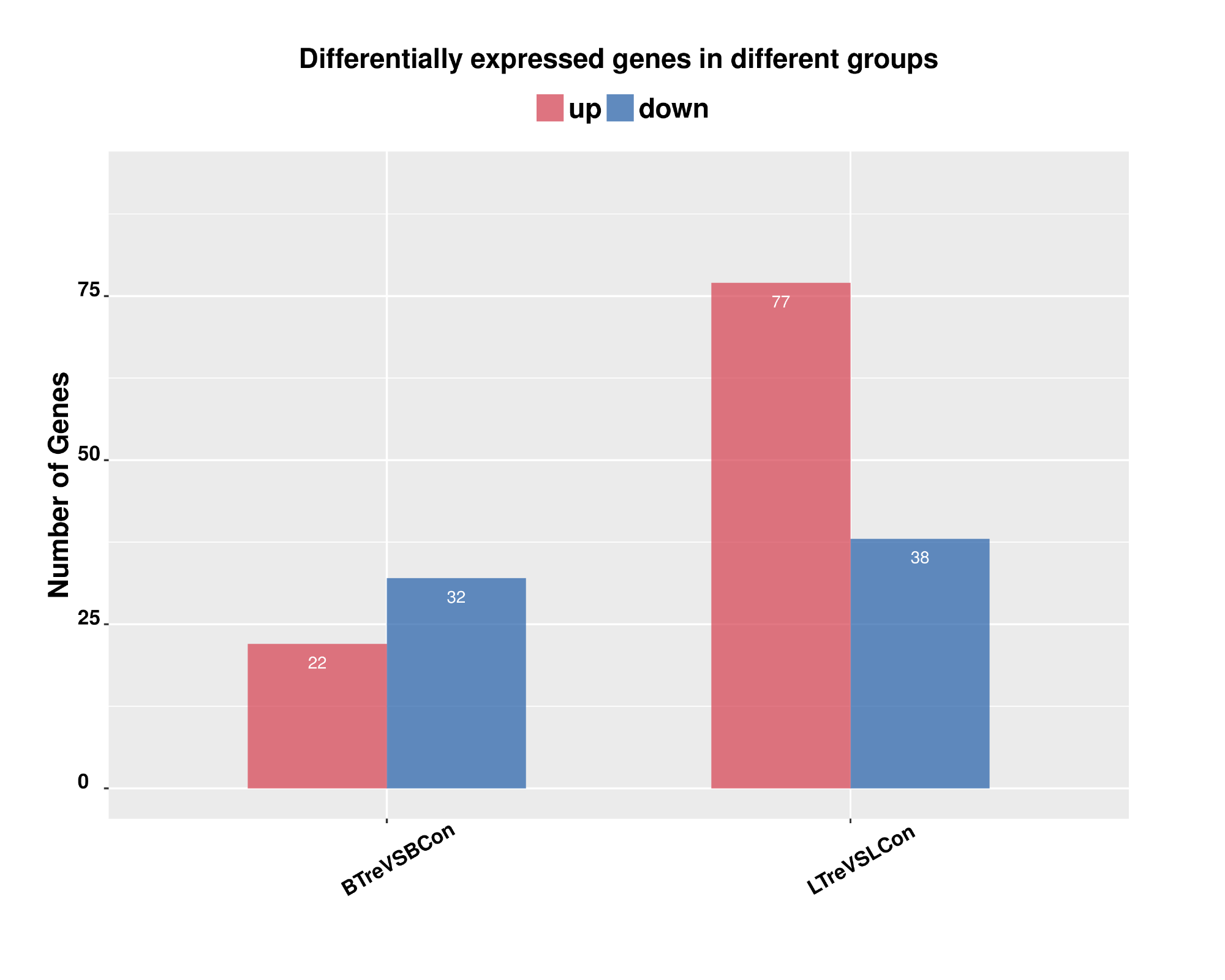
差異顯著差異表達基因上下調頻數統計柱狀圖用于統計差異基因數目
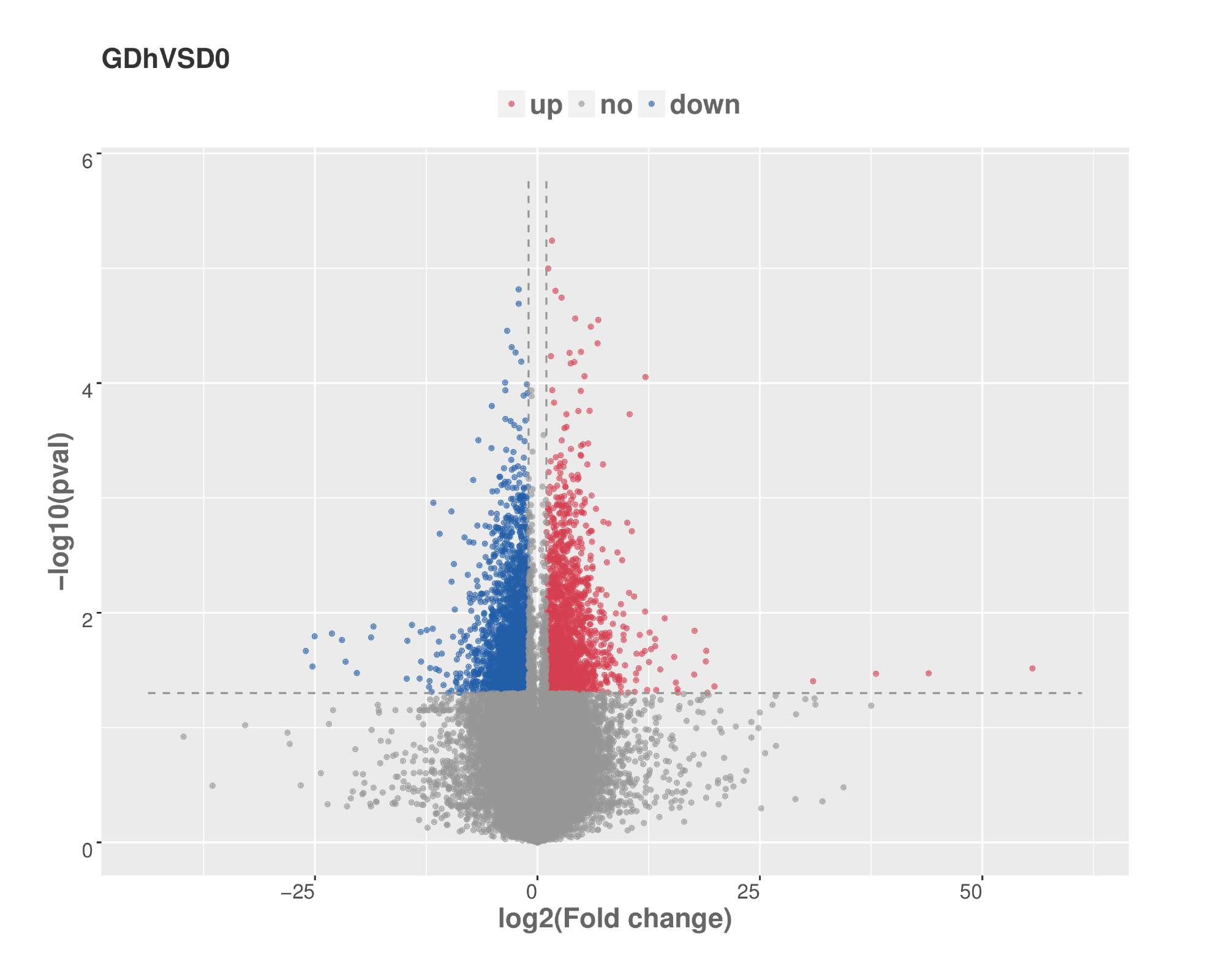
差異表達基因分析火山圖
用于了解差異表達基因的整體分布情況。以log2(foldchange)爲橫坐标,-log10(pvalue)爲縱坐标,對(duì)差異表達分析中所有的基因繪制火山圖。其中橫坐标代表基因在不同樣本中差異表達倍數變化;縱坐标代表基因表達量變化差異的統計學(xué)顯著性。
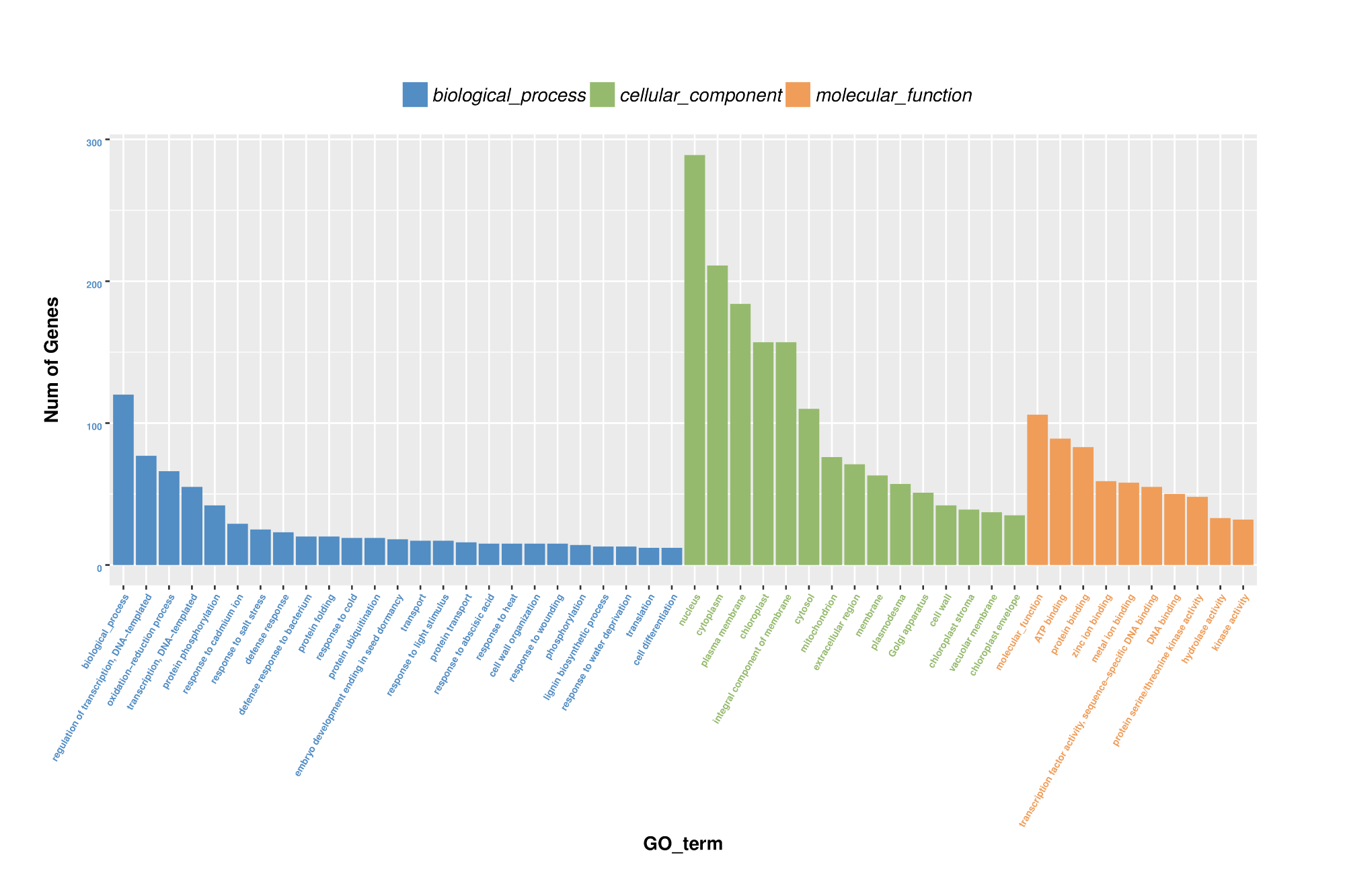
差異基因GO富集柱狀圖
用于反映在生物過(guò)程(biological process)、細胞組分(cellular component)和分子功能(néng)(molecular function)富集的GO term上差異基因的個數分布情況。
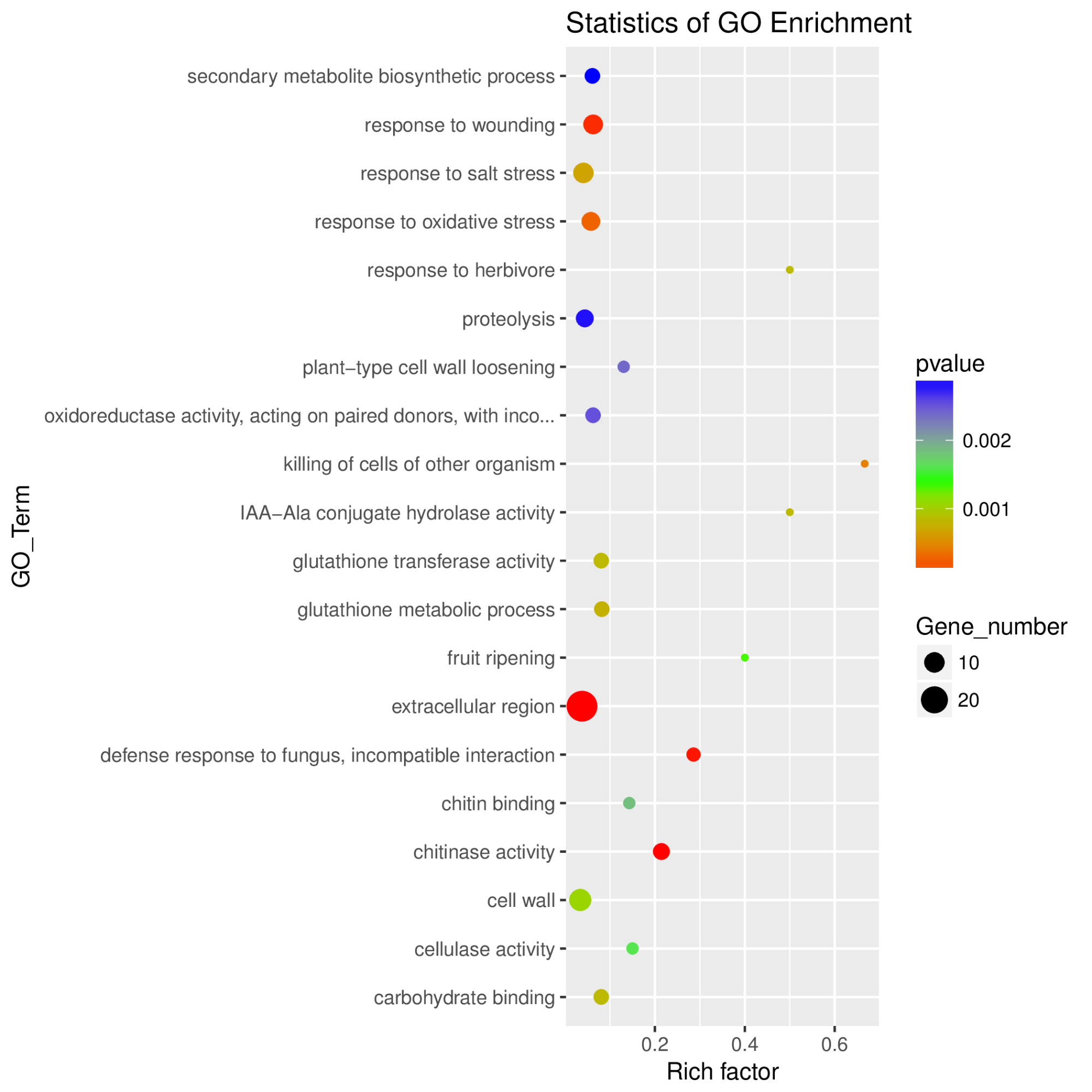
GO功能(néng)富集散點圖
用于展示GO term的富集情況。Rich factor表示位于該GO的差異基因個數/位于該GO的總基因數,Rich factor越大,GO富集程度越高。
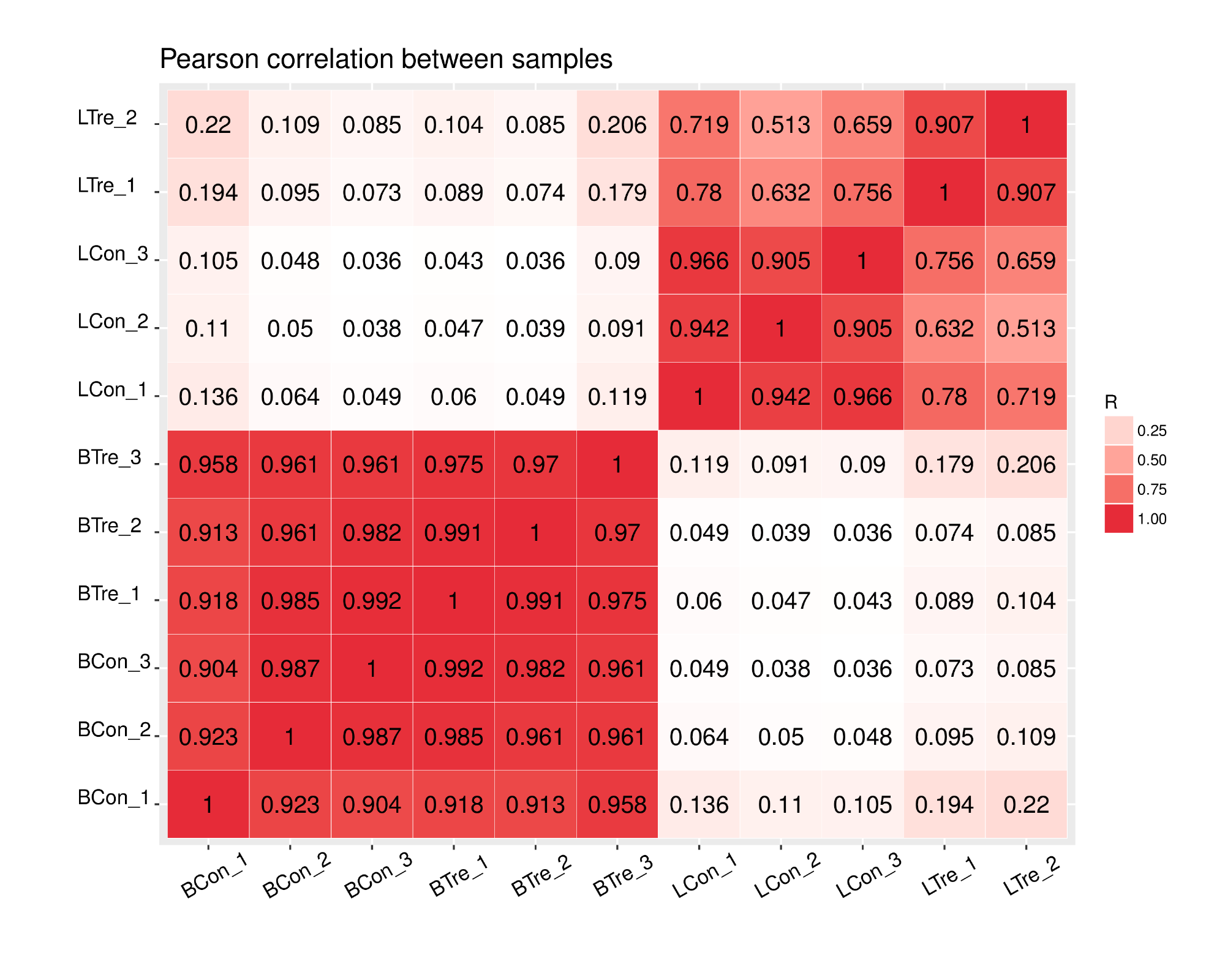
皮爾森相關系數圖
用于反映生物學(xué)重複、樣本相關性,确保後(hòu)續的差異基因分析得到更可靠的結果。
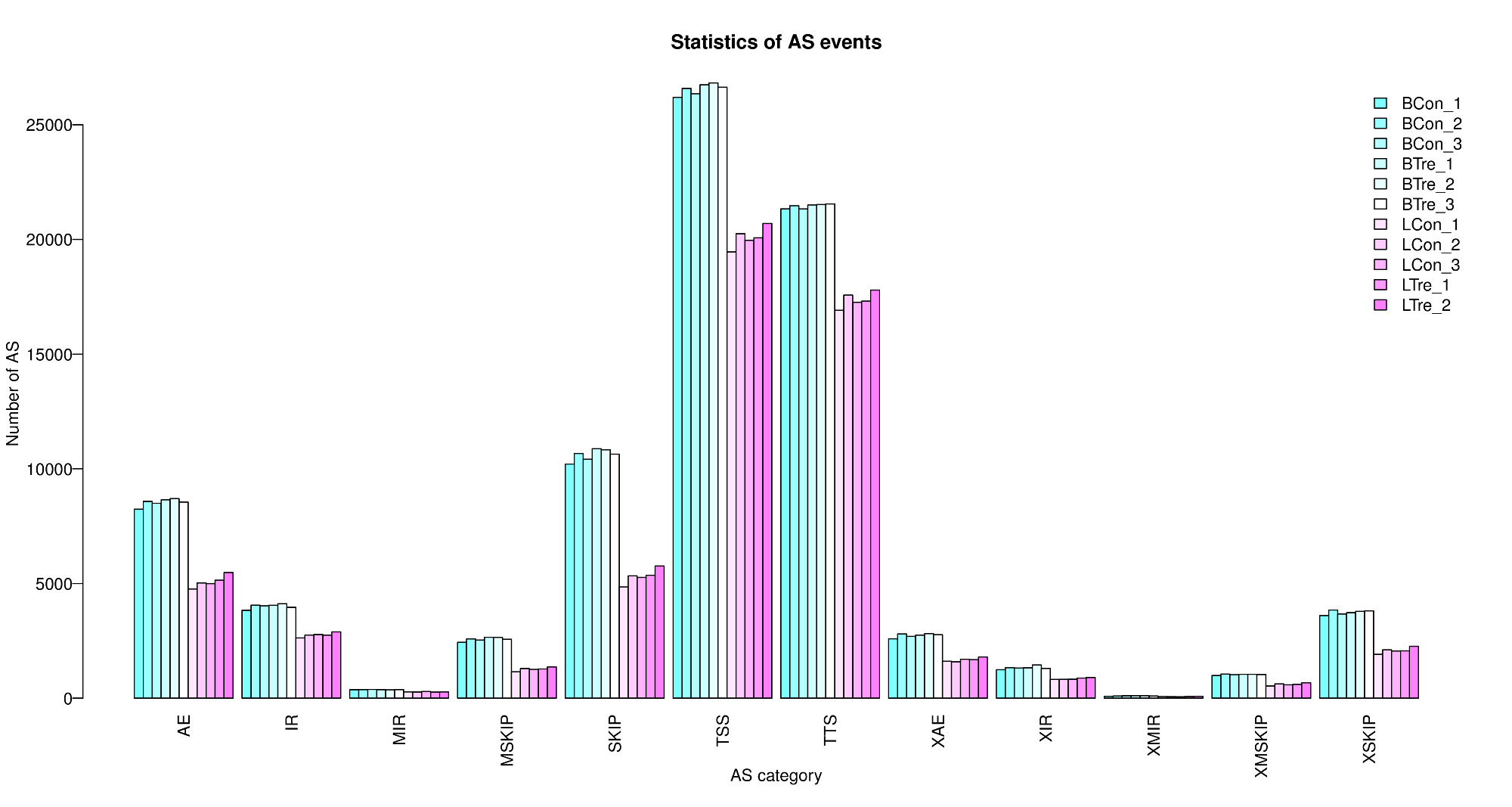
可變剪切統計圖
對(duì)該物種(zhǒng)及其相應的測序樣品進(jìn)行可變剪切事(shì)件的分類及統計。