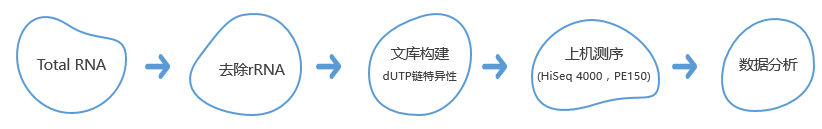
原核轉錄組測序是指利用高通量測序技術對(duì)原核生物的轉錄本(mRNA和非編碼RNA)進(jìn)行測序,全面(miàn)快速地獲取特定微生物在特定狀态下的所有轉錄本的信息。通過(guò)轉錄組測序不僅能(néng)夠對(duì)mRNA進(jìn)行表達定量分析,分析差異表達基因及其相應功能(néng),同時(shí)還(hái)能(néng)夠分析Non-coding RNA(sRNAs),揭示微生物不同表型形成(chéng)的分子調控機制和功能(néng)。 由于原核生物mRNA不具有polyA尾結構,需要采用去除rRNA的方法構建文庫,聯川生物針對(duì)不同的研究物種(zhǒng)采取有效的方法去除rRNA,保證數據質量。
技術優勢
磁珠法去除rRNA,rRNA去除效率好(hǎo),數據質量高
采用鏈特異性建庫,建庫穩定性好(hǎo)
除進(jìn)行mRNA定量分析外,還(hái)可以進(jìn)行反義轉錄本預測,基因結構分析等;
可以同時(shí)進(jìn)行mRNA和sRNA分析,sRNA 預測,變異分析等
技術路線
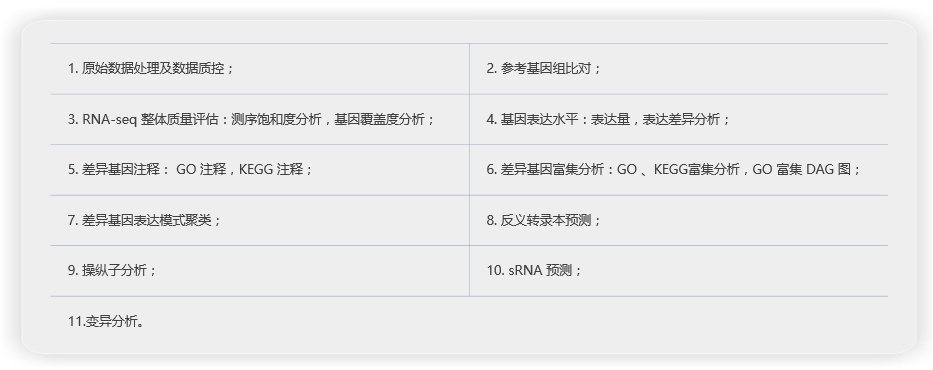
分析内容
樣本類型
微生物菌體(≥ 5×107),組織,環境樣品,總RNA等
建議總RNA起(qǐ)始量:≥3μg;濃度:≥70 ng/μL。
Q:原核生物與真核生物在進(jìn)行轉錄組測序文庫構建時(shí)有什麼(me)區别?A:在原核生物中,mRNA隻占全部RNA的1-5%,其餘絕大部分是核糖體RNA(rRNA),因此在開(kāi)展轉錄組測序前,需先將(jiāng)mRNA純化。然而,原核生物mRNA不具有polyA結構,無法直接利用oligoT將(jiāng)mRNA純化出來,如果直接用total RNA進(jìn)行測序,數據質量將(jiāng)非常差,大部分序列都(dōu)來自rRNA。
提高原核生物中mRNA的量,最主要的方式是去除total RNA中的rRNA。在建庫過(guò)程中顯加入磁珠將(jiāng)rRNA吸附,過(guò)濾掉磁珠後(hòu)的體系再進(jìn)行下遊建庫。
全基因組測序和轉錄組揭示藍藻對(duì)青藏高原極端氣候的适應機制
研究背景
青藏高原不僅是世界上最高且規模最大的高原,同時(shí)具有包括極大的溫差、低氧濃度、低壓、強紫外線輻射以及狂風等在内的極端環境,且有許多獨特的環境,包括雪山、鹽水湖泊及幹旱沙 漠等,具有極高的生物多樣性,爲研究适應性進(jìn)化提供了一個理想的天然實驗室。藍藻是最早的光合放氧生物,幾乎可以將(jiāng)所有緯度範圍的地區作爲栖息地,對(duì)于全球生态具有相當大的重要 性;藍藻可以适應可變滲透壓、持續低溫及強烈的紫外線輻射等環境。
研究結果
青藏高原藍藻基因組序列是 5.9 Mb,具有 39.2%的 G+C 含量,總共包括5362 個 CDS。系統進(jìn)化樹表明,這(zhè)一菌株屬于 Trichormus 和 Anabaena 集群。T. sp. NMC-1 和6個近 緣種(zhǒng)之間的基因組對(duì)比顯示,在 T. sp. NMC-1 基因組中,功能(néng)未知的基因占據了更高的比例(28.12%)。
轉錄組分析發(fā)現表達顯著上調的基因參與膜生物起(qǐ)源、翻譯、核糖體結構和生物起(qǐ)源、次生代謝産物生物合成(chéng)、氨基酸運輸和代謝、防禦機制等過(guò)程;相比之下,下調基因主要參與信 号轉導機制、膜生物起(qǐ)源及能(néng)源生産和轉換。進(jìn)一步的分析表明,CheY-like 基因、胞外多糖和類菌胞素氨基酸樣的氨基酸,可能(néng)在極端環境的适應性中扮演重要的角色。
研究結論
青藏高原引起(qǐ)極端環境而具有最高的生物多樣性,爲研究适應性進(jìn)化提供了一個理想的天然實驗室。該研究繪制了青藏高原藍藻的基因組序列草圖,并在低溫下進(jìn)行了全轉錄組測序,以探讨 T. sp. NMC-1 适應特殊環境的遺傳學(xué)機制。明顯正向(xiàng)選擇的、擴張純正群的和差異表達的一些基因參與信号轉導、細胞壁/膜生物起(qǐ)源、次生代謝産物的生物合成(chéng)、能(néng)源生産和轉換,旨在闡 述特殊的适應特征。這(zhè)些結果表明,藍藻對(duì)青藏高原極端環境的适應性背後(hòu),有著(zhe)複雜的遺傳學(xué)機制。
參考文獻
Qin Q, Huang Y, Ji Q, et al. The genome and transcriptome ofTrichormussp. NMC-1: insights into adaptation to extreme environments on the Qinghai-Tibet Plateau:[J]. Scientific Reports, 2016, 6:29404.
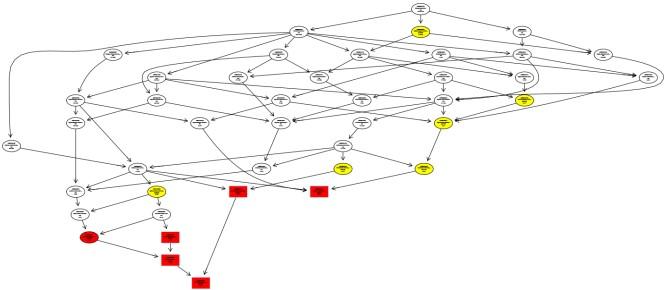
差異基因 GO 富集 DAG 圖有向(xiàng)無環圖(Directed Acyclic Graph, DAG)爲差異基因GO富集分析結果的圖形化展示方式。圖中的分支代表包含關系,從上至下所定義的功能(néng)範圍越來越小,一般選取GO富集分析的結果前10位作爲有向(xiàng)無環圖的主節點,并通過(guò)包含關系,將(jiāng)相關聯的GO Term一起(qǐ)展示,顔色的深淺代表富集程度。
差異基因表達模式聚類將(jiāng)有顯著差異的基因/轉錄本進(jìn)行表達模式聚類分析,采用距離計算算法: 樣本間爲spearman,基因間爲pearson,采用的聚類方法爲hcluster(complete算法)。
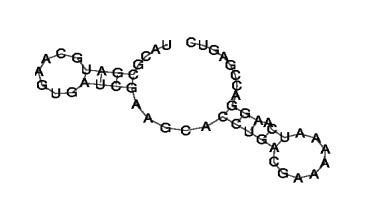
sRNA 預測原核生物中,長(cháng)度在 50~500nt 的非編碼 RNA 通常定義爲小 RNA(small RNA, sRNA)。用 Rockhopper 軟件 發(fā)現新轉錄本, 通過(guò)將(jiāng)其與 Uniport 數據庫進(jìn)行注釋,將(jiāng)未注釋到數據庫的轉錄本作爲潛在的非編碼 sRNA。用RNAFold 分析其莖環結構,進(jìn)行 二級結構預測。