

背景簡介
代謝組學(xué)的研究主要分爲非靶向(xiàng)代謝組學(xué)及靶向(xiàng)代謝組學(xué),非靶向(xiàng)代謝組學(xué)主要檢測樣品中代謝物的相對(duì)含量,而靶向(xiàng)代謝組學(xué)則是以标準品爲參照,對(duì)特定的代謝物群進(jìn)行有針對(duì)性地、特異性地檢測與分析,可驗證候選生物标記物并分析已知目标化合物。靶向(xiàng)代謝組分析通過(guò)對(duì)血液、尿液或其他體液以及組織中某一特定的代謝物的富集與準确定量定性分析,一方面(miàn)可以結合其它實驗數據揭示相關的分子生物學(xué)作用機制,另一方面(miàn),也可以爲後(hòu)續代謝分子标志物的深入研究和開(kāi)發(fā)利用提供有力支持。
技術優勢
高靈敏度:基于先進(jìn)的液相串聯質譜技術,可檢測到pg級化合物。
個性化定制: 靶向(xiàng)代謝組分析是針對(duì)特定代謝産物的分析,具有較強的特異性,可對(duì)不同性質代謝物建立不同的樣品
提取、富集與檢測方法。
低成(chéng)本:側重于相關特定組分特異性研究,篩檢目标代謝産物,檢測成(chéng)本較低。
跨組學(xué)分析:提供從基因組學(xué)、轉錄組學(xué)、蛋白質組學(xué)、到代謝組學(xué)的全程科技服務,深度剖析生物學(xué)現象。
技術路線
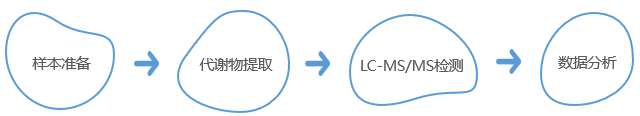
樣本類型
細胞,組織,尿液,全血,血清,血漿等
建議起(qǐ)始量(單次):血漿或血清>300 μL,尿液> 5 mL,組織> 100 mg,細胞>107個。
Q1:代謝組混合取樣的要求是怎樣的?
A:每份樣品混合來自至少3株以上的組織。以水稻葉片爲例:取同一時(shí)期,表型基本一緻的同一個部位的1片葉片,同時(shí)取6株,放到10ml離心管中,并迅速放入液氮中。
Q2:樣品重量和重複性問題怎麼(me)解決?
A:每份樣品取樣鮮重3-5g。準備3-6份生物學(xué)重複,代謝組通常做3個以上重複,剩餘備份。
案例一 水稻黃酮代謝模式和自然變異機制研究
黃酮是植物體中一類具有非常重要生物學(xué)功能(néng)的酚類化合物,目前,國(guó)内外研究者主要在拟南芥爲模式植物中研究,單子葉植物中研究很少。本研究以單子葉模式植物水稻爲研究材料,通過(guò)廣泛靶向(xiàng)代謝組技術測定不同水稻品種(zhǒng)、不同發(fā)育時(shí)期和不同組織材料中黃酮的含量。同時(shí),通過(guò)黃酮含量和黃酮合成(chéng)基因的序列多态性關聯分析,結果顯示:黃酮的自然變異與合成(chéng)基因的序列多态性相關。
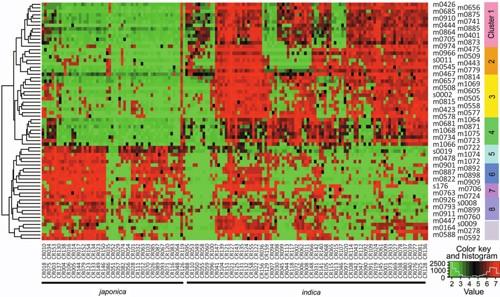
圖 水稻群體中黃酮物質的自然變異聚類熱圖
案例二 黃酮醇苯乙酰基轉移酶進(jìn)化分析揭示十字花科植物抗UV-B機制
植物具有避開(kāi)UV-B光照射的危害,但是精細的研究機理任然不清楚。在本研究中,通過(guò)測定拟南芥中代謝組研究了苯乙酰黃酮醇(saiginols)的自然變異規律和積累模式,尤其在拟南芥花器官中積累含量比較高。後(hòu)續通過(guò)測定一個亞群的拟南芥代謝組信息,尤其是位于北緯16°到43°地域的拟南芥品種(zhǒng)。基于LC-MS的代謝組檢測技術,共計檢測到了68種(zhǒng)代謝物,其中鑒定物質結構50種(zhǒng)(包括16種(zhǒng)硫代葡萄糖苷、3種(zhǒng)羟基肉桂酸、24種(zhǒng)黃酮化合物和7種(zhǒng)酚胺),未鑒定物質18種(zhǒng)。借助代謝組、轉錄組和IL材料重測序的信息,鑒定到FPT2基因家族參與合成(chéng)苯乙酰黃酮醇的途徑,研究進(jìn)一步發(fā)現苯乙酰黃酮醇的積累促進(jìn)植物抗UV-B的能(néng)力。
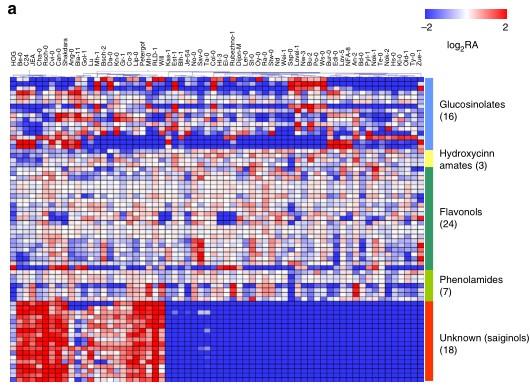
圖 拟南芥花器官材料次生代謝圖譜
參考文獻
1.Xuekui Dong, Wei Chen, Wensheng Wang . et al. Comprehensive profiling and natural variation of flavonoids in rice. Journal of Integrative Plant Biology, 2014, 56, 876-886.
2.Takayuki Tohge, Regina Wendenburg, Hirofumi Ishihara. et al. Characterization of a recently evolved flavonol-phenylacyltransferase gene provides signatures of natural light selection in Brassicaceae. Nature Communications, 2016.