背景簡介
iTRAQ ( Isobaric Tags for Relative and Absolute Quantitation) 技術是ABI公司推出的一項新的體外同位素标記技術。使用該技術可以對(duì)一個基因組表達的全部蛋白質或一個複雜的混合體系中的所有蛋白質進(jìn)行精确定量和鑒定。尋找差異表達蛋白,并揭示其細胞生理病理功能(néng)。聯川生物依托先進(jìn)的蛋白質譜分析平台、強大的信息分析和數據處理能(néng)力,爲用戶提供高效的蛋白質組鑒定與定量分析服務。
技術優勢
通量高:一次實驗即可對(duì)數千種(zhǒng)蛋白進(jìn)行定量分析,且最多可同時(shí)對(duì)8份樣本進(jìn)行定量分析
結果可靠:蛋白分析的靈敏度、特異性及重複性遠高于雙向(xiàng)電泳,并且定性與定量分析同步完成(chéng)
覆蓋廣泛:鑒定與定量分析樣本中表達的所有已知蛋白,包括高分子量蛋白、酸堿性蛋白、膜蛋白和不溶性蛋白等
支持關聯分析:綜合上遊轉錄組等實驗數據,提供多組學(xué)關聯分析
技術路線

分析内容

樣本類型
細胞,組織,尿液,全血,血清,血漿,總蛋白等
建議總蛋白起(qǐ)始量(單次):≥ 500 μg,濃度≥1μg/μL
用戶文章:
1.Wang L,et al. Proteome profiling reveals immune responses in Japanese flounder (Paralichthys olivaceus) infected with Edwardsiella tarda by iTRAQ analysis. Fish Shellfish Immunol. 2017 Jul;66:325-333. doi: 10.1016/j.fsi.2017.05.022
2.Zhang G, Zhang J, Wen X, et al. Comparative iTRAQ-based quantitative proteomic analysis of Pelteobagrus vachelli liver against acute hypoxia: Implications in metabolic responses.[J]. Proteomics, 2017.
3.Li R, Yu H, Yue Y, et al. Combined proteomics and transcriptomics identifies sting-related toxins of jellyfish Cyanea nozakii.[J]. Journal of Proteomics, 2016, 148:57.
4.Lv LX, et al.(2016) Integrated transcriptomic and proteomic analysis of the bile stress response in probiotic Lactobacillus salivarius LI01. Journal of Proteomics. doi.org/10.1016/j.jprot.2016.08.021
5.Lv LX, et al.(2016) Integrated transcriptomic and proteomic analysis of the bile stress response in probiotic Lactobacillus salivarius LI01. Journal of Proteomics. doi.org/10.1016/j.jprot.2016.08.021
6.Wang C, Liu C, Wei L, Shi L, Pan Z, Mao L, Wan X, Ping Z, Jiang T, Chen Z. (2016) A Group of Novel Serum Diagnostic Biomarkers for Multidrug-Resistant Tuberculosis by iTRAQ-2D LC-MS/MS and Solexa Sequencing. International Journal of Biological Sciences 12(2), 246-256.
7.Cui Y, et al. (2016) Transcriptomic/proteomic identification of allergens in the mite Tyrophagus putrescentiae. Allergy.DOI:10.1111/all.12999
8.Cao JY, et al. (2016) TMT-based quantitative proteomics analyses reveal novel defense mechanisms of B
Q1:iTRAQ能(néng)否鑒定新蛋白?
A:iTRAQ不能(néng)鑒定新蛋白,隻能(néng)做定量。定量的基本原理是根據肽段上報告基團信号的強弱來計算,不涉及内參蛋白。同時(shí),不能(néng)做未知蛋白鑒定。iTRAQ産品的分析都(dōu)是基于對(duì)數據庫的篩查,所以必須是序列已知的蛋白才可以檢測到。
Q2:iTRAQ蛋白定量能(néng)鑒定多少個蛋白,爲什麼(me)有些物種(zhǒng)鑒定到蛋白那麼(me)少?
A:對(duì)于可鑒定蛋白的數量确實因物種(zhǒng)有差異,原因是鑒定蛋白是通過(guò)篩查蛋白質數據庫來進(jìn)行的,如果某個物種(zhǒng)沒(méi)有很好(hǎo)的基因組注釋信息或轉錄組注釋信息,篩查得到的信息也會(huì)很少。尤其是植物類、昆蟲類、水産品類非模式物種(zhǒng),往往隻能(néng)鑒定不到1k個蛋白。
Q3:蛋白鑒定是不是一定要依賴于數據庫?
A:是的,所有蛋白産品裡(lǐ)的蛋白鑒定,都(dōu)是需要依賴數據庫的。數據庫信息的完整性,將(jiāng)對(duì)蛋白鑒定的結果産生一定影響。
Q4:iTRAQ結果的後(hòu)期驗證?
A:一般選擇注釋結果與所關注的生物過(guò)程緊密相關的蛋白或者差異顯著的蛋白來做後(hòu)期驗證。iTRAQ的後(hòu)期驗證,通常采用western blot。而植物中,可用抗體較少,通常的處理方式是不驗證或是自行制備多抗進(jìn)行驗證;亦可進(jìn)行mRNA的定量qPCR驗證,找mRNA和蛋白的關聯。兩(liǎng)種(zhǒng)方式都(dōu)已有文章發(fā)表。
轉錄組及蛋白質組學(xué)技術分析益生菌唾液乳杆菌LI01的膽汁應激反應
研究背景
最近發(fā)現幾種(zhǒng)唾液乳杆菌菌株,表現出良好(hǎo)的益生菌性質,如抗菌活性,炎症調節,甚至是腫瘤性病變減少等。從健康人體内分離的唾液乳杆菌LI01已經(jīng)在預防和治療肝衰竭中表現出了益生菌性質。對(duì)膽汁的耐受對(duì)于乳杆菌在胃腸道(dào)中的存活和發(fā)揮其益處至關重要。
研究結果
通過(guò)轉錄組測序和iTRAQ蛋白質組定量分析後(hòu)發(fā)現,與空白對(duì)照相比,添加ox膽汁溶液培養的LI01菌株中檢測到591個差異表達的基因和347個差異表達的蛋白質。利用GO和KEGG分析,發(fā)現差異表達的基因主要參與脅迫應答,糖酵解和轉運以及諸如碳水化合物代謝和氨基酸生物合成(chéng)等途徑;差異表達的蛋白質主要參與DNA修飾、基因表達和細胞組分降解和代謝過(guò)程的調節。
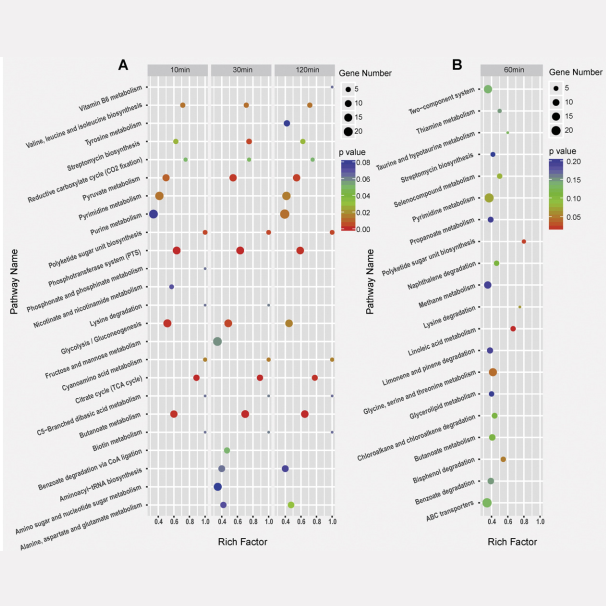
圖 差異表達基因及差異表達蛋白質的KEGG富集分析
研究還(hái)發(fā)現,LI01的膽汁耐受能(néng)力是基于高度重塑的細胞包膜以及增強的膽汁外排系統,而不是基于膽鹽水解酶的活性。一些差異表達的基因是與調節系統即應激反應和中樞代謝過(guò)程相關的基因,也在膽汁誘導的損傷預防和能(néng)量效率中起(qǐ)作用。此外,膽汁鹽似乎增強了LI01的蛋白水解和氨基酸攝取(特别是芳香族氨基酸)能(néng)力,這(zhè)個結果表明該菌株具有肝保護的性質。
總而言之,本研究的開(kāi)展建立了唾液乳杆菌LI01中膽汁應激的全局反應機制的模型。
參考文獻
Lv LX, et al.(2016) Integrated transcriptomic and proteomic analysis of the bile stress response in probiotic Lactobacillus salivarius LI01.Journal of Proteomics. doi.org/10.1016/j.jprot.2016.08.021
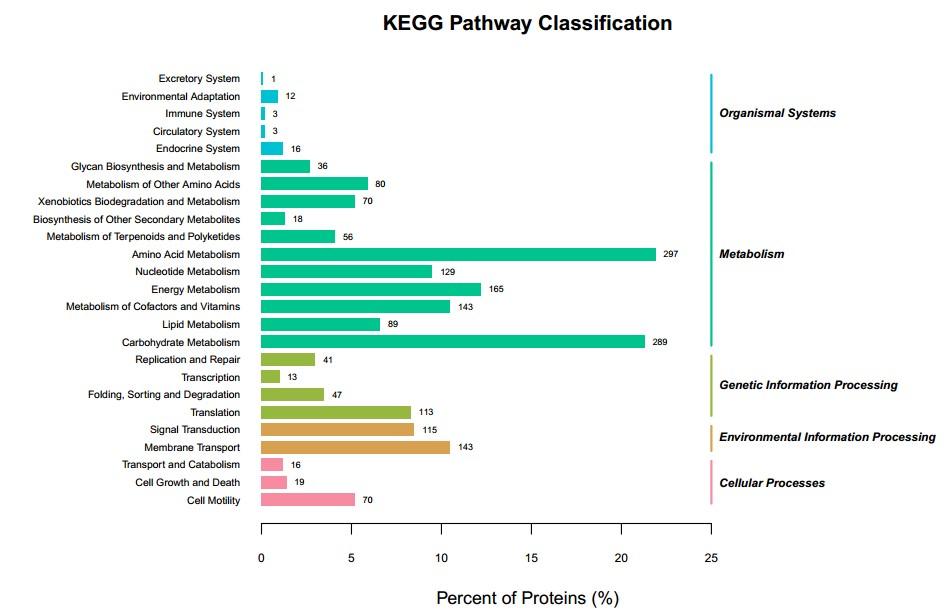
KEGG注釋根據KEGG注釋總表,可以對(duì)注釋到KEGG的差異蛋白總體情況進(jìn)行Unigene數目的統計。
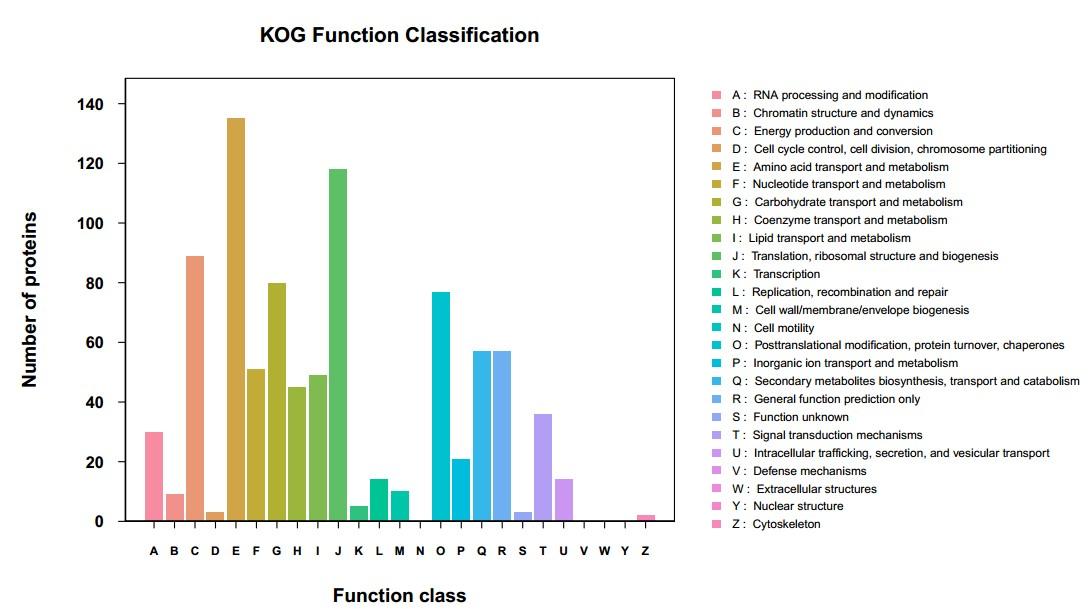
KOG/COG注釋COG(Clusters of Orthologous Groups of proteins),即同源蛋白簇。一般原核 生物用COG,真核生物用KOG。COG注釋作用:1. 通過(guò)已知蛋白對(duì)未知序列進(jìn)行功能(néng)注釋;2. 通過(guò)查看指定的COG編号對(duì)應的protein數目,存在及缺失,從而能(néng)推導特定的代謝途徑是否存在; 3. 每個COG編号是一類蛋白,將(jiāng)query序列和比對(duì)上的COG編号的proteins 進(jìn)行多序列比對(duì),能(néng)确定保守位點,分析其進(jìn)化關系。
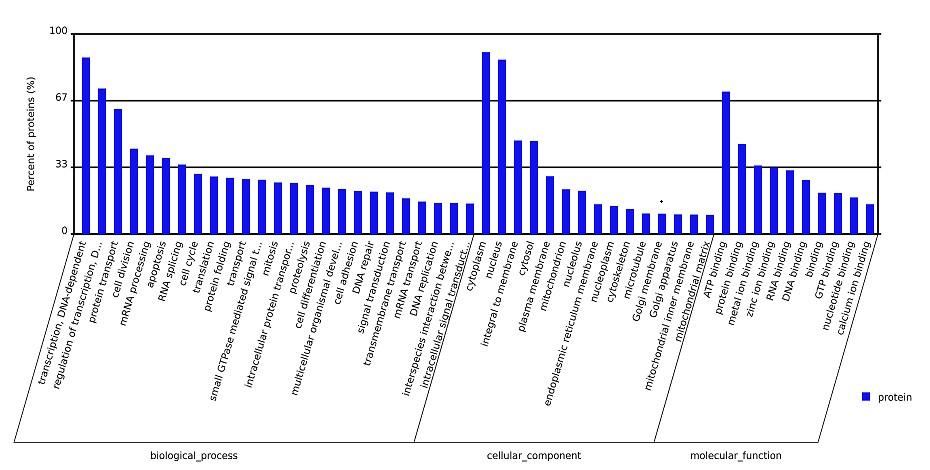
GO富集分析GO分爲三個功能(néng):生物進(jìn)程(biological process) 細胞組成(chéng)(cellular component)分子功能(néng)(molecular function)。一般進(jìn)行分析時(shí),會(huì) 取前50個差異較爲明顯的蛋白繪制GO注釋圖。分别對(duì)應這(zhè)三個功能(néng)的差 異蛋白數量爲25個,15個以及10個。
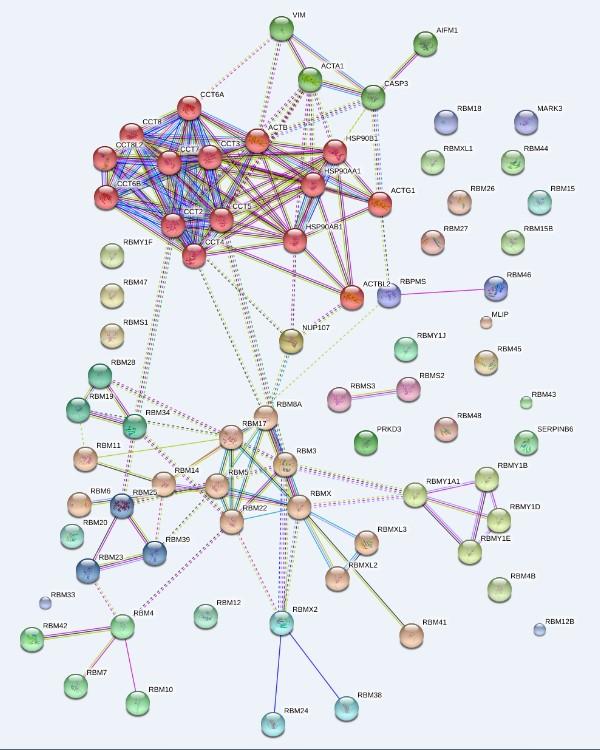
蛋白質network網絡構建蛋白與蛋白的互作關系一般用String軟件繪制,String可以用來查找蛋白間的作用關系及作用強度,同時(shí)也可以查看單個蛋白與其他相關聯的上下遊關系,可以爲老師研究蛋白互作關系提供思路 。