

背景簡介
宏基因組測序(Metagenomics Sequencing)是對(duì)環境樣品中的微生物群落的基因組進(jìn)行高通量測序,主要研究微生物種(zhǒng)群結構、基因功能(néng)活性、微生物之間的相互協作關系以及微生物與環境之間的關系。宏基因組測序研究擺脫了微生物分離純培養的限制,擴展了微生物資源的利用空間,爲環境微生物群落的研究提供了有效工具。
技術優勢
直接對(duì)特定環境中全部微生物群體基因組進(jìn)行測序。
環境微生物通常以群落方式共存和互作,宏基因組研究比單個個體研究更能(néng)反映微生物的真實生存狀态。
除分析微生物群體多樣性和豐度外,還(hái)可以解析微生物基因功能(néng)和參與的代謝通路,發(fā)掘新的具有特定功能(néng)的基因。
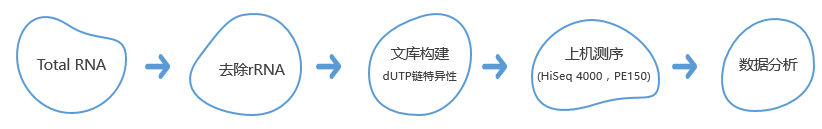
技術路線
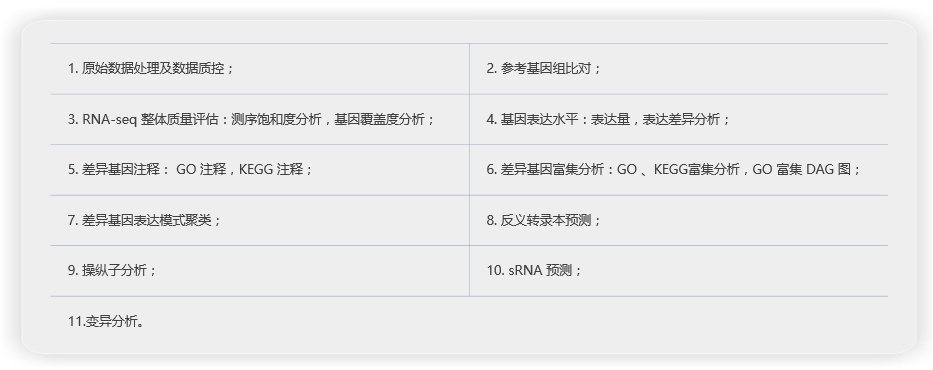
分析内容
樣本類型
環境樣品,總DNA等
建議總DNA起(qǐ)始量:5 μg,最低0.5 μg,濃度≥30 ng/μL(Qubit定量)
Q1:微生物群落研究方法比較?
A:16S rDNA測序:由于研究對(duì)象隻爲細菌的16S rDNA,因此16S測序技術更多隻用于研究群落物種(zhǒng)信息,也就(jiù)是利用OTU物種(zhǒng)分類、α和β多樣性分析等手段解答群落有什麼(me)物種(zhǒng),物種(zhǒng)關系是什麼(me)等問題。因此,這(zhè)種(zhǒng)技術更多地隻能(néng)了解到環境對(duì)微生物的組成(chéng)有何影響,更偏向(xiàng)于單向(xiàng)關系研究。
宏基因組測序:宏基因組的研究對(duì)象爲群落所有DNA,因此研究範圍更廣。理論上不單隻可以了解群落的組成(chéng)和多樣性等物種(zhǒng)信息,同時(shí),利用基因的注釋信息,還(hái)可以挖掘群落的核心功能(néng)和通路信息,在基因組層面(miàn)了解這(zhè)個群落到底有什麼(me)物種(zhǒng),這(zhè)些物種(zhǒng)能(néng)夠發(fā)揮什麼(me)功能(néng)。隻有了解群落功能(néng),才能(néng)知道(dào)它們對(duì)環境有什麼(me)影響,這(zhè)有利于進(jìn)行微生物與環境的雙向(xiàng)研究。
Q2:宏基因組測序原理?
A:將(jiāng)基因組DNA随機打斷成(chéng)若幹條500bp左右的小片段(類似于拼圖中的單個形狀不一的模塊),然後(hòu)連接接頭,在片段兩(liǎng)端加通用引物進(jìn)行PCR擴增測序。將(jiāng)reads進(jìn)行組裝拼接(類似于將(jiāng)衆多模塊拼成(chéng)一副完整圖片),得到基因序列,衆多基因構成(chéng)完整的基因集。同時(shí)將(jiāng)獲得的reads片段或組裝好(hǎo)的基因序列與NCBI數據庫進(jìn)行比對(duì),得到物種(zhǒng)注釋結果。
Q3:土壤樣本準備注意事(shì)項?
A:取土壤表層樣品(0~20 cm ),除去石塊和植物殘根等雜物,放入無菌封口袋中,立即置于冰盒 (建議使用幹冰,以利更好(hǎo)地保持微生物原貌)中運回實驗室 ,馬上進(jìn)行 DNA抽提。DNA 可置于 -20 ℃保存,長(cháng)期保存建議置于 -80℃存放。 如無法進(jìn)行DNA抽提,請盡快將(jiāng)樣本置于液氮中冷凍4小時(shí)以上,再轉移至-80℃保存 。
如采集根際土壤,拔取植株後(hòu),需抖落植株根部的大塊松散土壤,用已消毒的軟毛刷下附著(zhe)在植物根際的土壤,放入無菌封口袋中,立即置于冰盒中運回實驗室 ,馬上進(jìn)行DNA抽提。
Q4:水體樣品準備注意事(shì)項?
A:使用采水器采集水樣(建議1~2 L),置于冰盒(建議使用幹冰,以利更好(hǎo)地保持微生物原貌)中運回實驗室,立即用無菌的0.22 µm醋酸纖維濾膜抽濾水樣(若水體渾濁,可使用無菌的大孔膜預濾後(hòu)再使用 0.22 µm 濾膜;如 1張 0.22 µm濾膜無法收集全部菌體,可使用多張濾膜 ), 取出濾膜後(hòu)馬上進(jìn)行樣品DNA抽提。 DNA可置于-20 ℃保存,長(cháng)期保存建議置于-80℃存放 。如無法進(jìn)行DNA抽提,請盡快將(jiāng)樣本置于液氮中冷凍 4小時(shí)以上,再轉移至-80℃保存 。
Q5:糞便樣本準備注意事(shì)項?
A:采集新鮮糞便樣品并迅速置于無菌離心管中 ,置于冰盒(建議使用幹冰,以利更好(hǎo)地(建議使用幹冰,以利更好(hǎo)地保持微生物原貌)中運回實驗室,馬上進(jìn)行DNA抽提。DNA可置于-20 ℃保存,長(cháng)期保存建議置于-80℃存放。如無法進(jìn)行DNA抽提,請盡快將(jiāng)樣本置于液氮中冷凍4小時(shí)以上,再轉移至-80℃保存 。
宏基因組測序研究廢水處理過(guò)程中淤泥微生物抗性基因及移動元件
本文利用16S、宏基因組等技術研究在升溫厭氧消化處理污水的0d-57d天内,淤泥中抗性基因、移動元件以及抗性基因宿主微生物的變化。在0d時(shí),共發(fā)現13類抗性基因(Antibiotic Resistance Genes, ARG),其中9種(zhǒng)含量都(dōu)超過(guò)1ppm,ARG 總量從125.97ppm(0d)降低至50.65ppm(57d),其中氯黴素抗性基因、多耐藥性抗性基因等抗性基因降低多達50%以上。高溫處理後(hòu),移動元件總量由528.73ppm降低至391.36ppm。
本文研究表明,在消化處理淤泥過(guò)程中,随溫度升高,會(huì)明顯降低淤泥中的抗性基因、移動基因組元件以及抗性基因宿主微生物的含量,其中抗性基因的降低可能(néng)是與移動元件以及宿主微生物的降低有關,因此高溫厭氧消化處理城市廢水污泥能(néng)夠有效的抑制抗性基因擴散到環境中。

參考文獻
Tian Z, Zhang Y, Yu B, et al. Changes of resistome, mobilome and potential hosts of antibiotic resistance genes during the transformation of anaerobic digestion from mesophilic to thermophilic.[J]. Water Research, 2016, 98:261.
樣品聚類分析通過(guò)對(duì)樣品進(jìn)行聚類分析,構建聚類樹,可以研究不同樣品之間的相似性。Bray-Curtis 距離是系統聚類法中使用最普遍的一個距離指标,它主要用來刻畫樣品間的相近程度,它的大小是進(jìn)行樣品分類的主要依據。根據各物種(zhǒng)在各樣品中的豐度表出發(fā),以Bray-Curtis距離矩陣進(jìn)行樣品間聚類分 析,并將(jiāng)聚類結果與各樣品的物種(zhǒng)相對(duì)豐度整合進(jìn)行展示。

KEGG注釋根據KEGG注釋總表,可以對(duì)KEGG注釋的總體情況進(jìn)行Unigene數目的統計.

eggNOG分類統計圖eggNOG可以分爲四個層次。第一層包括:1.信息存儲和加工;2.細胞過(guò)程和信号轉導; 3.新陳代謝;4.未确定分類。第二層進(jìn)一步細分成(chéng)25個分類,每一個分類都(dōu)可以用一個字母表示。第三層爲共同功能(néng)描述(Consensus Functional Description)。第四層爲具體的直系同源蛋白簇。將(jiāng)Unigenes與eggNOG庫的蛋白序列進(jìn)行比對(duì)(blastp,evalue≤1e-5),取分值最高的eggNOG比對(duì)結果序列(Hits)的注釋作爲該Unigene序列的注釋。結合eggNOG數據庫的層次結構,可以統計出eggNOG各個層級或水平的信息。

CAZy柱狀圖CAZy是一個特殊的數據庫,緻力于分析碳水化合物活性酶的基因組,結構和生 信息等。CAZy 數據庫主要涵蓋 6 大功能(néng)類:糖苷水解酶,糖基轉移酶,多糖裂合酶,碳水化合物酯酶,輔助氧化還(hái) 原酶和碳水化合物結合模塊。6個功能(néng)大類又可以進(jìn)一步分成(chéng)各功能(néng)小類,整個數據庫具有明顯的兩(liǎng)個層次的結構。在過(guò)去的幾年裡(lǐ),CAZy分類家族不 斷擴展,爲基因組和宏基因組的分析提供更完備的數據。
